Построено филогенетическое дерево средневековой чумной палочки
В ходе второй пандемии чумы бактерия Yersinia pestis терроризировала Европу и окрестные регионы с 14 по 18 века нашей эры и привела к гибели (по разным оценкам) от 30 до 60 процентов населения Европы. Сегодня ученым удалось восстановить ход событий и определить, какие изменения происходили в геноме чумной палочки в зависимости от места и времени вспышки болезни.
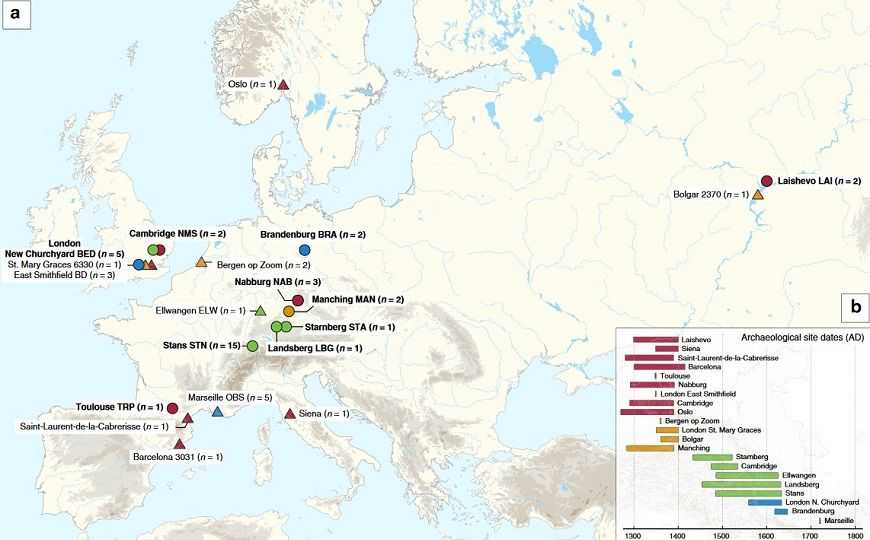
a. Карта, показывающая географическое расположение археологических памятников, из которых были реконструированы геномы Y. pestis (XIV-XVIII века н.э.) геномов. Количество геномов (n), полученных из каждого сайта, указано в скобках. Местоположения ранее опубликованных геномов отмечены треугольниками, тогда как геномы, которые описаны в этом исследовании, показаны кругами (метки выделены жирным шрифтом). B. Хронологии образцов, объединяющие археологические и радиоуглеродные данные ранее опубликованных и новых изолятов пандемии второй чумы.
Credit: Maria A. Spyrou et al. Распространяется по Creative Commons Attribution 4.0 International License (CC BY 4.0)
Международная группа исследователей проанализировала останки людей времен второй пандемии чумы из девяти археологических памятников, расположенных в Англии, Франции, Германии, Швейцарии и России. По образцам ДНК из зубов жертв чумы реконструировали 34 древних генома Y. pestis. Сначала образцы проверяли на наличие специфичного для чумной палочки гена pla (активатор плазминогена), используя количественную ПЦР. Затем, чтобы секвенировать бактериальный геном отдельно от генома носителя, было применено полногеномное секвенирование с обогащением в растворе. Для этого метода создают библиотеку ДНК образца, в состав которой входят как бактериальная, так и человеческая ДНК. К ней в раствор добавляют специально синтезированную биотинилированную библиотеку из молекул РНК, комплементарных молекулам ДНК, которые необходимо секвенировать. В растворе наживка специфически связывается с нужной ДНК и с помощью биотина оседает на покрытых стрептавидином шариках. Лишняя ДНК образца вымывается при смене раствора, а для оставшегося материала проводят секвенирование нового поколения.
На основе полученных данных исследователи построили филогенетическое дерево для Y. pestis, используя метод максимального правдоподобия. Оказалось, что Черная Смерть, вспышка чумы 1346–1353 годов, с которой началась пандемия, всегда была ассоциирована с одним и тем же штаммом чумной палочки; штаммы из останков тех, кто заболевал чумой вне вспышки, имели незначительные генетические отличия.
Анализ геномов чумной палочки после Черной Смерти показал, что линия Y. pestis разделилась на множество генетически различных ветвей. Это позволило чуме использовать в качестве резервуаров более широкий спектр живых организмов. Кроме того, полученные данные подтверждают, что чума впервые пришла в Европу с востока: штамм, оказавшийся предком всех последующих, был обнаружен в Татарстане, в городе Лаишево.
Также оказалось, что со временем штаммы чумной палочки, ответственные за поздние стадии пандемии, утрачивают область генома, содержащую в том числе и часть вирулентных генов. Такая же делеция присутствовала и у штаммов, связанных с первой пандемией чумы (541–750 н.э.). Это позволило исследователям сделать вывод о том, что в ходе обеих пандемий эволюция бактерии Y. pestis шла примерно одинаково.
"Учитывая, что данная делеция произошла в эволюционных линиях, обе из которых вымерли, влияние этих генов на способность чумной палочки использовать людей и блох в качестве резервуаров может быть важной темой для изучения в будущем," — считает Кирстен Бос, лидер исследовательской группы и одна из авторов статьи.
Источники
Maria A. Spyrou et al. // Phylogeography of the second plague pandemic revealed through analysis of historical Yersinia pestis genomes // Nature Communications, 2019; DOI: 10.1038/s41467-019-12154-0
Meredith L. Carpenter et al. // Pulling out the 1%: Whole-Genome Capture for the Targeted Enrichment of Ancient DNA Sequencing Libraries // The American Journal of Human Genetics, 2013; DOI: 10.1016/j.ajhg.2013.10.002


 Меню
Меню





 Все темы
Все темы





 0
0