Транспозаза цианобактерий, направляемая гидРНК, делает вставки в геном
Новый инструмент для геномного редактирования — CRISPR-ассоциированная транспозаза цианобактерии Scytonema hofmanni — эффективно и точно встраивает ДНК в таргетный сайт. Авторы работы — сотрудники Института Брода под руководством Фэна Чжана, а также Евгений Кунин и Кира Макарова из Национального центра биотехнологической информации.
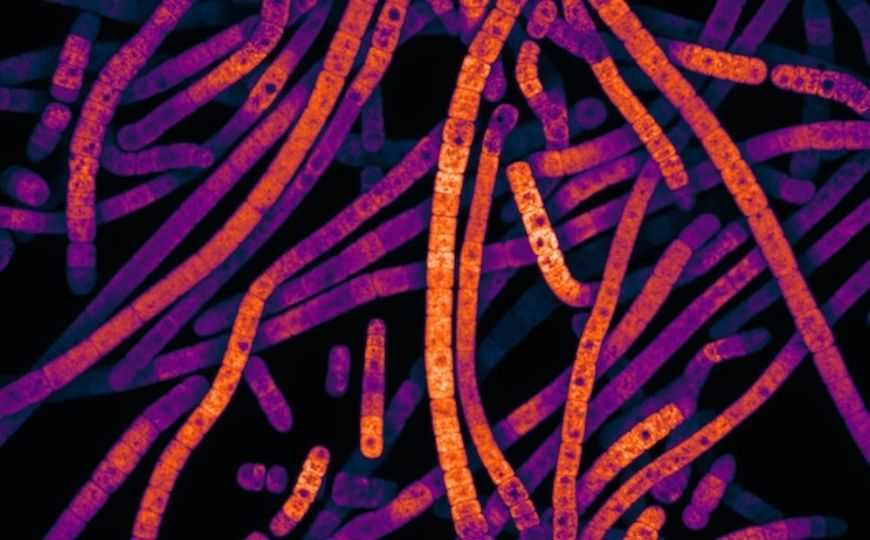
Флуоресцентная микрофотография цианобактерий Scytonema hofmanni
Fei Chen | http://news.mit.edu/2019/gene-editing-system-precisely-inserts-large-dna-sequences-0612
Системы CRISPR-Cas, применяемые для редактирования генома, как правило, вносят разрывы в определенный участок ДНК. (Особая тема — редакторы азотистых оснований, не вносящие разрывы, но они имеют свои ограничения.) Затем разрывы восстанавливаются клеточными системами репарации, по пути гомологичной рекомбинации либо негомологичного соединения концов, однако их эффективность зависит от типа клетки и в любом случае далека от 100%-ной. Поэтому методики, использующие Cas9, хорошо подходит для повреждения генов, которые по тем или иным причинам необходимо инактивировать. Сложнее добиться замены поврежденного участка правильным или встройки фрагмента в место разрыва. Оказалось, что последнюю задачу можно решить с помощью самодостаточных механизмов встраивания ДНК, таких как транспозазы.
Исследователи из Института Брода и Гарварда (МТИ, США) и Национального центра биотехнологической информации (Национальные институты здравоохранения США) описали инструмент для внесения вставок в выбранные участки ДНК — CRISPR-ассоциированную транспозазу цианобактерии Scytonema hofmanni.
Недавние исследования, проведенные в Национальном центре биотехнологической информации под руководством Евгения Кунина, выявили в геномах микроорганизмов ассоциации между Tn7-подобными транспозонами и системами CRISPR-Cas подтипов I-F, I-B, V-K. Это говорило о том, что транспозоны могли «похитить» эффекторы CRISPR (в случае системы V-K CRISPR эффектором является белок Cas12k, ранее известный как C2c5). Это позволило бы создавать R-петли, то есть трехспиральные комплексы ДНК и РНК, в таргетных сайтах, и облегчило бы распространение транспозонов посредством плазмид и фагов.
Авторы новой работы, опубликованной в Science, предположили, что Tn7-подобную транспозазу можно направить к таргетным сайтам с помощью гидРНК (gRNA) и репрограммировать так, чтобы вставлять фрагменты ДНК в определенные участки генома.
Для эксперимента было выбрано два Tn7-подобных транспозона, кодирующих CRISPR-Cas (CRISPR-associated transposase, CAST) системы подтипа V-K; этот подтип встречается у цианобактерий. Выбранные локусы CAST длиной 20–25 килобаз содержали ген Tn7-подобной транспозазы на одном конце и ген Cas12k на другом. Исследователи убедились, что CAST, полученный от цианобактерии S. hofmanni (его назвали ShCAST), может вносить разрезы в различные последовательности ДНК.
Чтобы проверить, можно ли превратить ShCAST в инструмент направленной коррекции ДНК, выбрали 48 локусов в геноме E. coli и внедрили в бактерии плазмиды с генами ShCAST и генами гидРНК к этим локусам. ПЦР-анализ показал, что инсерции произошли в 29 локусах из 48 (это 60,4%), а цифровая капельная ПЦР подтвердила рост доли инсерций до 80% спустя 16 часов после начала эксперимента.
«Взятые вместе, эти результаты говорят о том, что ShCAST эффективно и точно встраивает ДНК в таргетный сайт, — пишут авторы. Однако необходимы дальнейшие эксперименты для понимания функции каждой субъединицы транспозазы в комплексе CAST и дальнейшая белковая инженерия компонентов транспозазы, чтобы увеличить специфичность таргетинга.
Источник
Jonathan Strecker et al. // RNA-guided DNA insertion with CRISPR-associated transposases // Science, 2019,DOI: 10.1126/science.aax9181








 Меню
Меню





 Все темы
Все темы





 0
0