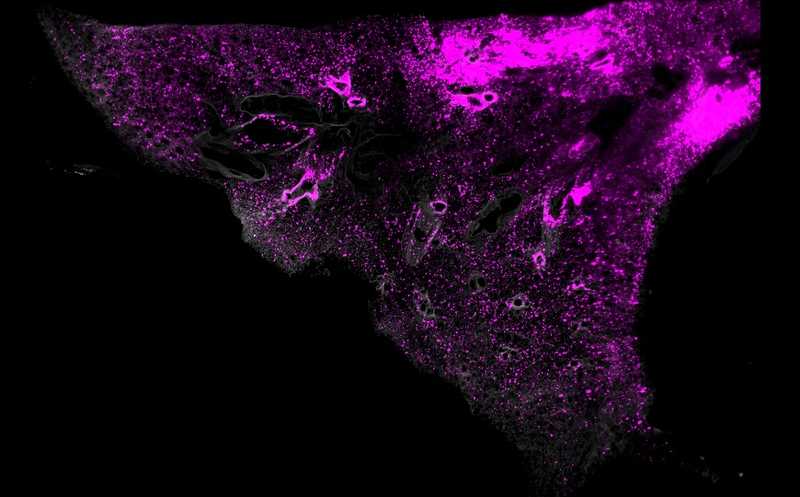
Оценить здоровье колонии пчел позволит ДНК из мёда
Ученые исследовали образцы меда с греческой пасеки в течение трех сезонов. Они показали, что ДНК из меда не только позволяет узнать, пыльцой и нектаром каких растений питались пчелы, но и выявить состав микробиоты кишечника пчелы и присутствие паразитов. Авторы показали, что пищевые привычки пчел зависят от сезона, в то время как микробиота остается на удивление неизменной.

Пчелы выполняют очень важную роль в качестве опылителей. Но в настоящее время эти насекомые сталкиваются с такими стрессорами, как изменение климата, снижение биоразнообразия пищевых ресурсов, урбанизация, патогены, гербициды и инсектициды. Идет поиск методов, которые позволили бы оценить здоровье пчелиной колонии. Все большее значение приобретают геномные технологии, в частности, секвенирование ДНК меда. Это неинвазивный подход, который позволяет детектировать многие виды, с которыми взаимодействуют пчелы. Такую ДНК называют «ДНК из окружающей среды» (environmental DNA, eDNA), для ее определения используют «метагеномное исследование с помощью шотган-секвенирования» (shotgun metagenomics).
Метод был разработан недавно, но ученые из Греции, авторы нового исследования, значительно его оптимизировали. В первую очередь они улучшили методы экстракции eDNA из образцов меда и анализа их метагеномного состава. После этого они собирали образцы на греческой пасеке в течение трех сезонов и оценивали, как изменялась eDNA меда.
Авторы успешно идентифицировали растения, нектаром и пыльцой которых питались пчелы. Результаты секвенирования подтвердили in silico, а также с помощью визуального анализа пыльцы и изучения растений, произрастающих в районе пасеки. Всего авторы идентифицировали 40 видов растений, которые отображают биоразнообразие окружающей пасеку флоры. Состав растительной eDNA значительно изменялся в образцах, собранных в разные сезоны.
Но большая часть ДНК в образцах меда принадлежала не растениям, а микробиоте кишечника пчел. Преобладала ДНК Lactobacillus kunkeii, также авторы отметили следы сезонной оппортунистической бактерии Spiroplasma melliferum. Состав микробитоты кишечника отличался постоянством в разных образцах.
Таким образом, анализ меда позволяет охарактеризовать как патогены пчел, так и их коровый микробиом. Важно, что метод может выявить заражение колонии паразитическим клещем Varroa destructor, что, по мнению авторов, поможет в будущих широкомасштабных исследованиях.
Источник
Galanis A., et al. Bee foraging preferences, microbiota and pathogens revealed by direct shotgun metagenomics of honey // Molecular Ecology Resources (2022), published May 20, 2022, DOI: 10.1111/1755-0998.13626


 Меню
Меню





 Все темы
Все темы



 0
0