Комплексный анализ экзома быстро выявляет генетические аномалии плода
В Нидерландах в течение года тестировали новые протоколы для поиска генетических вариантов, объясняющих нарушения развития, выявленные на УЗИ. Новые методы позволяют проводить комплексный поиск вариаций числа копий и однонуклеотидных вариантов по результатам полноэкзомного секвенирования. С таким подходом анализировать образцы можно быстрее и дешевле.
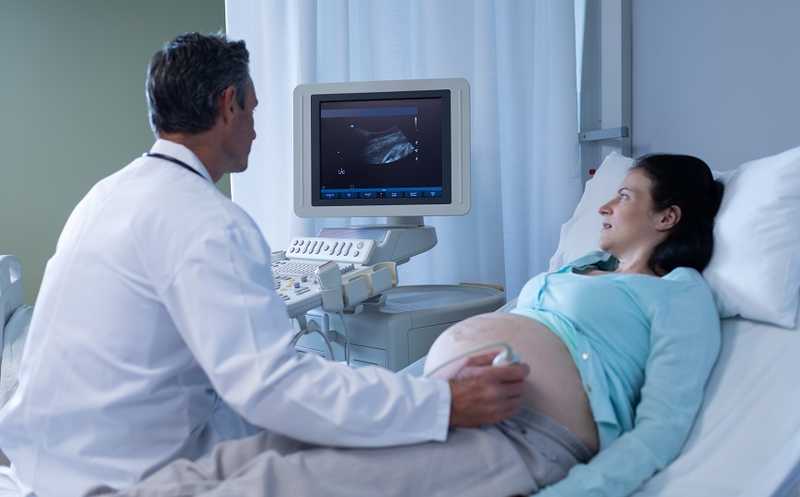
Нарушения развития плода на УЗИ выявляются в 2–3% беременностей. В большинстве случаев они обусловлены генетическими аномалиями: анеуплоидиями, вариациями числа копий (CNV), унипарентальными (однородительскими) дисомиями (UPD) или однонуклеотидными вариантами (SNV). Для обнаружения подобных нарушений используют различные методы генетического анализа. Анализ анеуплоидий проводится преимущественно при помощи качественной флуоресцентной ПЦР (QF-PCR); CNV выявляются ДНК-микрочипами; SNV — анализом экзома. Однако необходимость проведения отдельного анализа для каждого типа аномалий (часто последовательно) приводит к увеличению денежных и временных затрат. Лаборатории на базе Университета Радбауд в Неймегене и Маастрихтского университета (Нидерланды) в течении года тестировали в клинических условиях новый протокол для комплексного анализа экзома на CNV и SNV.
Протоколы тестировали с декабря 2020 по декабрь 2021 года. Авторы анализировали образцы ворсинок хориона (CV) или амниотической жидкости (AF). В первую очередь проводился QF-PCR поиск наиболее часто встречающихся анеуплоидий. В случаях, если нарушение количества хромосом обнаружено не было или оно не объясняло дефекты, выявленные при УЗИ, родителям предлагали дальнейшее генетическое тестирование. По выбору пациента проводился либо только полноэкзомный анализ CNV (WES-CNV), либо комплексный анализ CNV и SNV (WES-CNV/SNV). В качестве алгоритма поиска CNV использовался CoNIFER v0.2.0. Для анализа Y хромосомы и делеций использовался алгоритм ExomeDepth v0.8.4. SNV-анализ проводился по существующим генетическим панелям с дальнейшей возможностью полноэкзомного поиска вариантов. По запросу пациента после неинформативного WES-CNV анализа проводилось также SNV-исследование по уже полученным результатам секвенирования.
За описанный период было отобрано 415 инвазивных образцов (138 CV и 277 AF). В 93 случаях были обнаружены аномалии при тестировании QF-PCR (49,3% образцов CV, 9% образцов AF).
Из 70 образцов CV с неинформативным результатом QF-PCR для 52 был запрошен тест WES-CNV. В четырех случаях этот анализ выявил генетические причины нарушений развития. Для двух из 48 оставшихся образцов были выявлены CNV, унаследованные от родителя без явных нарушений. Как следствие, данные вариации были признаны доброкачественными. Для 20 образцов было запрошено дополнительное SNV-тестирование. В десяти случаях оно выявило генетические вариации, потенциально являющиеся причиной нарушений развития.
Для 16 образцов CV было запрошено тестирование WES-CNV/SNV. Были обнаружены пять SNV и одно CNV-нарушение, объясняющие нарушения развития плода.
Для образцов AF информативный результат QF-PCR был получен в 25 случаях. WES-CNV анализ был запрошен в 108 случаях. Он выявил пять CNV, потенциально объясняющих нарушения развития. Дополнительный SNV-анализ был запрошен в 17 случаях. Для трех образцов были выявлены потенциально патогенные SNV.
Анализ WES-CNV/SNV образцов AF был запрошен в 132 случаях. Было обнаружено 17 CNV и 24 SNV, объясняющих нарушения развития. Более высокий процент обнаружения генетических аномалий в образцах AF по сравнению с CV, вероятнее всего, объясняется тем, что забор AF обычно происходит при более серьезных нарушений развития.
В протокол тестирования также была включена проверка на контаминацию. В качестве допустимого предела было установлено значение в 5%. Контаминация более 5% была выявлена только в одном образце. Во всех случаях обнаруженная контаминация обуславливалась клетками матери.
Время на получение результатов (TAT) замерялось как период от забора образца до получения итоговых результатов. Для QF-PCR TAT составляло 1–5 дней. Среднее TAT для WES-CNV составило 14 дней; для WES-CNV/SNV — 13 дней. Дополнительный SNV-анализ образцов WES-CNV проводился в среднем за 6–7 дней. В случаях, когда дополнительный анализ проводился в непосредственной близости от границы возраста плода, после которой в Нидерландах запрещено проведение аборта, и результаты были нужны срочно, их удавалось получить за 3–4 дня. В отдельных случаях результаты дополнительного анализа были получены в течении одного рабочего дня.
По результатам годового тестирования новых протоколов ученые сообщают, что процент выявления патогенных генетических вариантов не отличался от традиционных методов. Тем не менее, группа сообщает об определенных преимущества новых методов. Так, сокращение времени на получение результатов обеспечивает возможность более ранней консультации родителей по результатам генетического анализа. Возможность проведения дополнительных анализов на уже полученных результатах секвенирования экзома позволяет значительно сократить денежные затраты. Стоимость исследований может снизиться еще сильнее с дальнейшим развитием технологий секвенирования. Таким образом, использование анализов WES-CNV и WES-CNV/SNV может значительно повысить эффективность клинических исследований.
Трансплантация матки и ЭКО помогают при «безнадежных» случаях бесплодия
Источник:
Faas B.H.W., et al. All-in-one whole exome sequencing strategy with simultaneous CNV-, SNV- and Absence-of-Heterozygosity analysis in fetuses with structural ultrasound anomalies: A one year’s experience. // Prenatal Diagnosis, published January 17, 2023. DOI: 10.1002/pd.6314


 Меню
Меню





 Все темы
Все темы




 0
0