Ученые создали карту сходства белков, управляющих судьбой РНК в клетке
Использовав мультиомиксный подход, то есть результаты трех высокопроизводительных экспериментов различного типа, молекулярные биологи и биоинформатики показали, насколько было недооценено разнообразие ролей и функций РНК-связывающих белков в регуляции судьбы РНК в клетке от транскрипции до деградации. Важно, что выбор конкретной роли определяется как взаимодействиями белков друг с другом, так и множеством их РНК-мишеней.
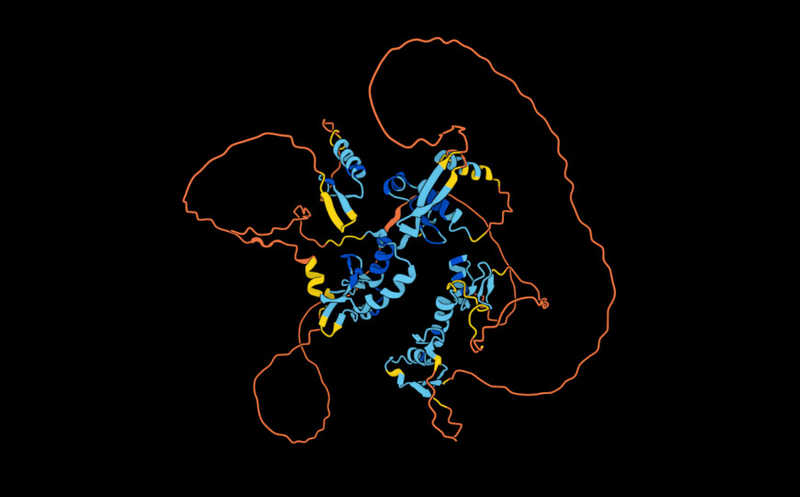
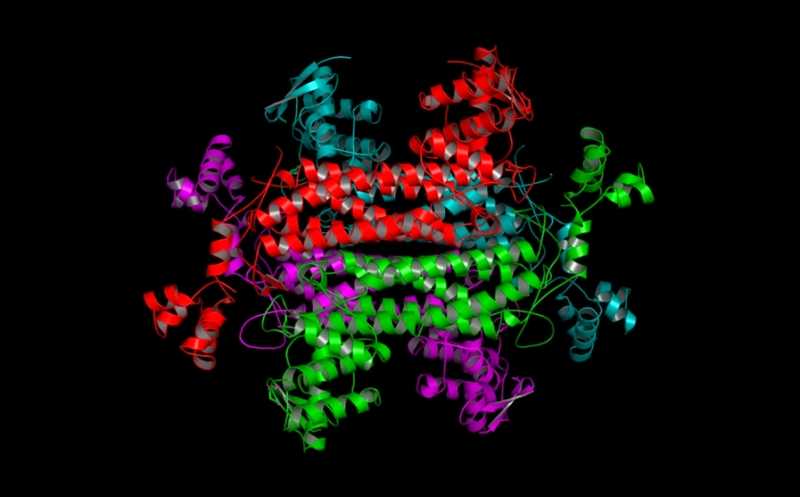
Структура человеческого ZNF800 — ранее не аннотированного РНК-связывающего белка, роль которого в транскрипции и формировании рибосом описали авторы обсуждаемой работы. Изображение получено с помощью AlphaFold2.
Credit:
alphafold.ebi.ac.uk | CC BY
Коллектив молекулярных биологов из Калифорнийского университета в Сан-Франциско и российских биоинформатиков из Института белка в Пущино и Института общей генетики (команда д.б.н. Ивана Кулаковского, ученика чл.-корр. РАН В.Ю. Макеева) исследовал работу РНК-связывающих белков (РСБ). Эти белки отвечают за регуляцию посттранскрипционных процессов, то есть происходящих уже после того, как с кодирующего участка ДНК — гена — синтезируется молекула РНК. Посттранскрипционные процессы включают в себя и сплайсинг, и выход РНК из ядра, и трансляцию мРНК, и распад РНК в клетке. В геноме человека вместе и по отдельности работают около полутора тысяч РСБ, при этом их функциональные особенности, а также характер взаимодействия с РНК и друг с другом еще недостаточно изучены.
«Тогда как изучение регуляции активности генов ДНК-распознающими факторами транскрипции является хоть и не тривиальной, но уже привычной задачей, для решения которой существуют популярные экспериментальные и вычислительные методы, исследование пост-транскрипционной регуляции существенно отстает и анализ РНК-связывающих белков по сей день остается проблемой для многих научных групп, — рассказал PCR.NEWS один из первых авторов исследования Андрей Буян. — Так, лишь у малого числа РСБ определены четкие паттерны их участков связывания, при этом функциональное разнообразие последствий РНК-белковых взаимодействий очень велико: они могут влиять на синтез РНК, ее созревание, транспорт, трансляцию, стабильность, или же на несколько этапов сразу».
Предметом исследования авторов статьи, опубликованной в журнале Nature Communications, стали около сотни РНК-связывающих белков. Для их изучения специалисты применили три подхода. Во-первых, они провели эксперимент по анализу белковых партнеров РСБ, для чего к РСБ присоединяли биотинлигазу — фермент, навешивающей биотиновую метку на рядом расположенные белковые молекулы. Таким образом выявляли белки, которые работают в одном комплексе. Ученые предполагали, что с разными партнерами один и тот же белок может выполнять разные функции.
Во-вторых, авторы посмотрели, что происходит с клеткой в отсутствие того или иного РСБ. Для этого использовали CRISPR конструкцию с гидовой РНК к данному белку. Последствия нокдауна белка они оценивали путем анализа транскриптомов единичных клеток. Сравнивая профиль клетки при отсутствии одного и другого белка, делали вывод о сходстве или несходстве их функциональности.
В третьем эксперименте оценивали взаимодействие РСБ с РНК-мишенью методами иммунопреципитации, когда белок выделяют при помощи антител к нему, а потом с использованием обратной транскрипции и технологии высокопроизводительного секвенирования определяют последовательности связанных с ним молекул РНК. В ходе этого эксперимента белки сравнивали уже по их РНК-мишеням.
«В каждом типе экспериментов мы оценивали сходство белков друг с другом, а затем объединяли результаты всех трех подходов, — объясняет Андрей Буян. — Если мы видели, что белки похожи по всем трем модальностям, значит, они входят в один функциональный кластер». Результатом работы стала мультимодальная карта для сотни РСБ, отражающая их близость друг к другу с точки зрения совместной работы и общих функций, и вовлеченность отдельных белков, их комплексов и более широких функциональных групп в те или иные биологические процессы.
При этом ученые подтвердили, что большинство РСБ одновременно относятся ко множеству регуляторных модулей. Например, есть белки, которые включены в трансляцию, но они же обеспечивают выход мРНК из ядра, они же регулируют ее стабильность в клетке, они же участвуют в сплайсинге. То есть они сначала помогли синтезировать РНК, а потом сопровождают ее на последующих этапах. «Видимо достаточно часто РНК-связывающие белки сопровождают свою мишень в разных клеточных процессах от транскрипции до деградации», — говорит Андрей Буян.
На следующем этапе работы специалисты подтвердили обнаруженные закономерности на четырех конкретных белках, про работу которых до этого было известно довольно мало. Например, для белка ZNF800, ранее не аннотированного, было показано, что он, с одной стороны, регулирует транскрипцию, а с другой стороны, вовлечен в формирование рибосом. Другой белок, ZC3H11A, участвующий в экспорте мРНК из ядра, как оказалось, также влияет на сплайсинг: в некоторых случаях он приводит к альтернативному сплайсингу своих РНК-мишеней. То же обнаружили для белка TAF15, который не только играет роль транскрипционного фактора, но и участвует в сплайсинге связываемых молекул РНК, а также влияет на их стабильность, трансляцию с этих молекул и даже на то, как долго такие РНК будут существовать в клетке. А для белка QKI, известного как фактор сплайсинга, обнаружилась также роль репрессора транскрипции — он способен регулировать экспрессию генов путем закрытия участков хроматина, а также связываться с промоторами своих мишеней.
Авторы работы пришли к выводу, что большинство РСБ мультифункциональны и играют различную роль в зависимости от контекста — это правило, а не исключение, причем контекст задают и белки-партнеры, и РНК-мишени, и клеточный компартмент. Ученые надеются, что полученные в этой работе экспериментальные данные и результаты помогут мировому сообществу в дальнейшем изучении и функциональной аннотации многообразия РНК-связывающих белков.
AlphaFold2 помог расшифровать взаимодействие транскрипционных активаторов и их кофакторов
Источник
Matvei Khoroshkin, et al. Systematic Identification of Post-Transcriptional Regulatory Modules. // Nature Communications, 2024, 15, Article number: 7872. DOI: 10.1038/s41467-024-52215-7


 Меню
Меню





 Все темы
Все темы



 0
0