BRB-seq: недорогое и высокопроизводительное секвенирование РНК
Новый подход к секвенированию РНК менее требователен к качеству матрицы, имеет производительность, сопоставимую с TruSeq, но при этом дешевле в 25 раз.
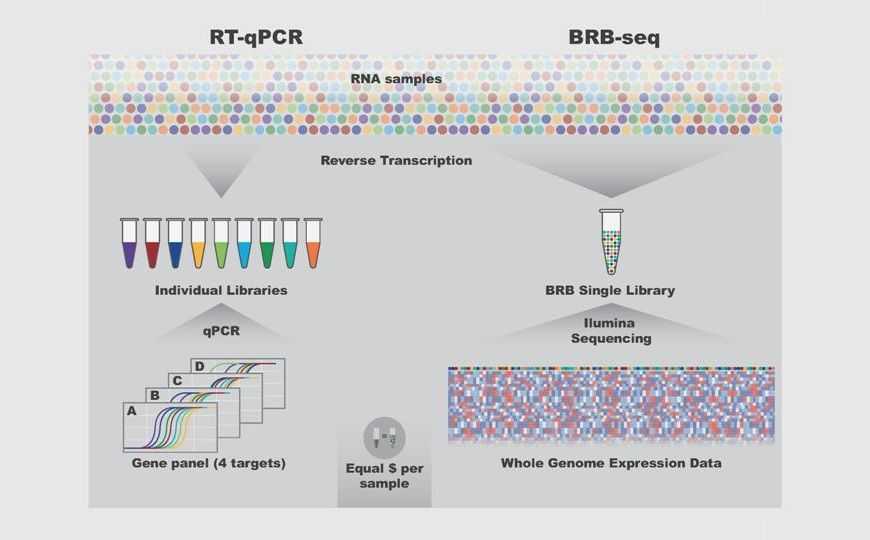
За последние годы цена на высокопроизводительное секвенирование снизилась. Тем не менее стоимость и сложность стандартной подготовки библиотек для секвенирования РНК все еще высоки, что не позволяет этому подходу стать таким же рутинным, как количественная ПЦР, особенно, когда необходимо проанализировать много образцов. Все методы получения библиотек для секвенирования РНК включают одни и те же этапы: обратную транскрипцию, фрагментацию, баркодирование и амплификацию. (Баркодирование — присоединение к каждому фрагменту баркода, технической последовательности, которая позволяет при обработке данных отнести прочтение к конкретному образцу. Это дает возможность мультиплексирования — объединения и одновременной обработки всех образцов, что снижает себестоимость.) Различия наблюдаются в последовательности проведения и тонкостях этих этапов.
Авторы статьи, опубликованной в Genome Biology, предлагают новый подход к секвенированию РНК — BRB-seq (Bulk RNA Barcoding and sequencing), позволяющий объединить преимущества различных методов РНК-секвенирования.
Традиционные методы массовой транскриптомики, например Illumina TruSeq Stranded mRNA, используют позднее мультиплексирование, при котором все образцы приходится обрабатывать отдельно. Для РНК-секвенирования большого количества образцов, в частности, для исследования РНК в единичных клетках (CEL-seq2, SCRB-seq, STRT-seq) используется принцип раннего мультиплексирования: баркодирование проводится во время реакции обратной транскрипции, это позволяет выполнять остальные этапы уже для всех образцов одновременно. Поскольку баркоды прикрепляются к транскриптам до фрагментации, считывания охватывают только концы транскриптов, а не полные последовательности, но для многих задач транскриптомики это приемлемо; как отмечают авторы, стандартные методы РНК-секвенирования генерируют больше информации, чем нужно.
В качестве экспериментальной основы был выбран подход SCRB-seq, как наиболее экономичный по времени и затратам. Сравнив его с Illumina TruSeq, авторы поняли, что в своем первоначальном виде SCRB-seq не может конкурировать с TruSeq из-за низкого качества секвенирования (большого количества нецелевых последовательностей в библиотеках и т.п.). Поэтому для BRB-seq они изменили ключевые этапы рабочего процесса, включая дизайн праймеров с баркодом, начальное количество РНК и количество циклов амплификации.
Авторы сравнили протокол BRB-seq с протоколом Illumina TruSeq Stranded mRNA при секвенировании одних и тех же образцов. Результаты подтвердили высокую общую производительность подхода BRB-seq, который показал сопоставимую эффективность и чувствительность по сравнению с TruSeq, но при этом стоимость процедуры была в десятки раз ниже.
По мнению авторов, новый подход позволит анализировать экспрессию генов в десятках образцов одновременно с минимальными усилиями и затратами. Один человек с помощью BRB-seq за день может подготовить до 192 образцов. Количество образцов в одной библиотеке масштабируется и зависит только от количества доступных баркодов и желаемого покрытия. Стоимость подготовки для одного образца составит около 2 долларов США при условии одновременной подготовки 96 образцов. Добавив стоимость секвенирования, авторы оценивают общие затраты на транскриптом примерно в 20 долларов. Основное ограничение BRB-seq — требование точной оценки количества РНК в образцах до обратной транскрипции, поскольку различия между образцами приведут к неравномерному распределению считываний последовательностей.


 Меню
Меню





 Все темы
Все темы



 0
0