Корейские ученые перепутали редактор оснований, который модифицировали
Сверхкомпактный редактор аденинов, о создании которого сообщили корейские ученые в 2022 году, сделан не из транспозазы TnpB, а из хорошо известной нуклеазы Un1Cas12f1. Это установили группы под руководством Виргиниюса Шикшниса и Дженнифер Дудны, которые исследовали локусы, кодирующие тот и другой фермент.
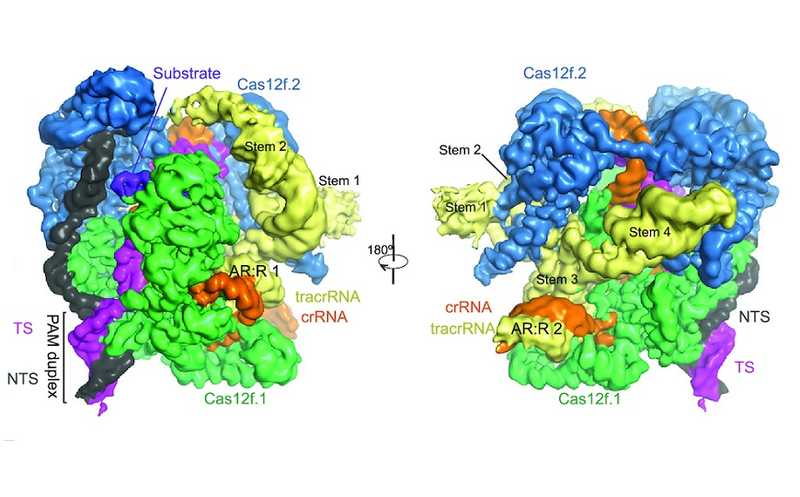
Структура Cas12f в комплексе с гидРНК и мишенью
Credit:
Nucleic Acids Research, 2021, DOI: 10.1093/nar/gkab179
В августе 2022 года исследователи Корейского исследовательского института бионаук и биотехнологий (KRIBB) опубликовали статью о сверхкомпактном редакторе адениновых оснований, который упаковывается в аденоассоциированный вектор (см. обзор Георгия Чистова на PCR.NEWS). Другие редакторы аденинов в AAV не помещаются, таким образом, новая разработка представлялась важным шагом к доставке редактирующей конструкции в организм. Авторы утверждали, что в конструкции используется модифицированная транспозаза TnpB. Технологию модификации назвали TaRGET (Tiny nuclease RNA-based Genome Editing Technology): эта модификация несколько удлиняла белковую молекулу, но она все же умещалась в вирус, а эффективность редактирования выросла значительно. (Изначально фермент был получен из генома археи и в эукариотической клетке работал неидеально.)
На этой неделе в Nature Chemical Biology вышли комментарии Виргиниюса Шикшниса и Таутвидаса Карвелиса из Вильнюсского университета, а также Дженнифер Дудны с коллегами. Классики метода CRISPR-редактирования привлекают внимание к неловкой ситуации: объектом генноинженерных усилий корейской команды оказалась известная ранее нуклеаза Un1Cas12f1, а не TnpB. «Хотя белки TnpB обладают биотехнологическим потенциалом в качестве эндонуклеаз, управляемых РНК, важно определить, какие белки действительно являются TnpB, а какие представляют собой Cas12, чтобы избежать путаницы», — замечают Дудна с коллегами.
Исследование истории эволюции систем CRISPR класса II показало, что РНК-управляемые эндонуклеазы Cas9 и Cas12 произошли от белков IscB и TnpB, которые кодируются транспозонами семейства IS200/IS605. Гомология между белками, теперь известными как IscB и TnpB, и эффекторами CRISPR класса II была отмечена еще в 2014 году, а в 2021 году выяснилось, что IscB и TnpB кодируют РНК-управляемые эндонуклеазы. Корейские исследователи предполагали, что разработали свои миниатюрные редакторы оснований на базе TnpB. Но это оказался фермент Cas12, который включен в системе CRISPR, а не TnpB, происходящий из транспозонов.
Надо заметить, что ранее корейцы использовали инженерную гид РНК, чтобы повысить эффективность редактирования того самого Un1Cas12f1 (также известного как Cas14a.1) — этот фермент тоже происходит из архей и тоже отличается низкой активностью в эукариотических клетках. Он принадлежит к кладе Cas12f, тесно связанной с TnpB.
В новой работе исследователи из KRIBB обратили внимание, что TnpB, полученная от архей группы Candidatus woesearchaeota, имеет последовательность, идентичную Un1Cas12f1, за исключением дополнительных 28 аминокислотных остатков на N-конце. Они убедились, что сконструированная ими гидРНК для Un1Cas12f1 повышает эффективность редактирования с помощью TnpB.
Однако анализ генных последовательностей показывает, что «TnpB» на самом деле является настоящим Cas12f1. Существуют значительные различия между локусами, кодирующими Cas12, и локусами, кодирующими TnpB. Команды Дудны и Шикшниса изучили локус, приведенный в работе корейских исследователей, где находился предполагаемый TnpB, и убедились, что он идентичен локусу, кодирующему Un1Cas12f1. В том числе идеально совпали сиквенсы локализованного там же массива CRISPR, участков генов cas2 и cas4, гена «tnpB» (то есть cas12f1) и примыкающих к ним некодирующих последовательностей. А вот элементы, характерные для транспозонов, в этом локусе отсутствуют. Дополнительные 28 аминокислот на N-конце возникли из-за трансляции с альтернативного кодона.
Таким образом, локус 'TnpB' — на самом деле последовательность, которую в 2018 году описали Дудна с коллегами для Un1Cas12f1, также в поисках компактной нуклеазы. «Любое различие между “TnpB” и Un1Cas12f1 является исключительно различием в аннотации и предсказании генов, а не различием в биологической функции», — подчеркивают Дудна и соавторы.
Исследователи из KRIBB внесли в свою статью соответствующее исправление, теперь речь в ней идет о модификации Un1Cas12f1. По их мнению, номенклатурная проблема не снижает качества исследования, поскольку модифицированный ими вариант фермента демонстрирует существенное повышение активности, и предложенный ими способ модификации редакторов оснований имеет самостоятельную ценность.
Источники
Kim, D. Y. et al. Hypercompact adenine base editors based on transposase B guided by engineered RNA. Nature Chemical Biology. 18, 1005–1013. 2022. DOI: 10.1038/s41589-022-01077-5
Karvelis, T., Siksnys, V. Mis-annotation of TnpB: case of TaRGET-ABE. Nature Chemical Biology. 2023. DOI: 10.1038/s41589-022-01242-w
Yoon, P.H., Adler, B.A. & Doudna, J.A. To TnpB or not TnpB? Cas12 is the answer. Nature Chemical Biology. 2023. DOI: 10.1038/s41589-022-01243-9


 Меню
Меню





 Все темы
Все темы



 0
0