МД-2023: Российский нанопоровый секвенатор и хранение больших данных
Продолжение секции «Новейшие разработки в области массового параллельного секвенирования (NGS)» было не менее интересным. Как хранить огромные объемы данных после секвенирования, можно ли с помощью генетического анализа выяснить потенциал спортсмена, как обстоит дело с секвенированием в Зимбабве и, наконец, каков он — российский нанопоровый секвенатор? Читайте в этом материале.

Вторая часть секции началась с того, что Тонни Савади Декстер (Tonny Savadye Deckster), PhD, National Biotechnology Authority (NBA), выступил с докладом «The use of sequence-based molecular techniques to detect human and animal disease, the case of COVID-19 Diagnosis in Zimbabwe».
Пандемия COVID-19 подтолкнула развитие генетических технологий в Зимбабве.
Первый случай коронавирусной инфекции был зафиксирован в марте 2020 года. Сначала в стране не было способов молекулярной диагностики, заболевание идентифицировали по симптомам и по результатам клинического осмотра.
Нужны были способы лабораторного тестирования. RT-ПЦР была доступна только в частных клиниках и очень дорого стоила. Потом появились более дешевые антигенные тесты, которые были доступны государственным лабораториям.
Впоследствии амплификаторы поставили и в государственные лаборатории, быстро научили людей работать с ними. Тестирование стало дешевле.
С ростом числа случаев пришлось автоматизировать процесс. Антигенные тесты использовали для скрининга, а ПЦР — для подтверждения диагноза. Серологические методы были менее популярными.
Нужда в секвенировании образцов была связана с возникновением новых вариантов и необходимостью в мониторинге.
В апреле 2021 года у Thermo Fisher закупили платформу для секвенирования нового поколения (Genexus). Был организован геномный центр. Все образцы направляли в национальный биобанк для хранения и секвенирования.

Сейчас в стране есть несколько специалистов, которые могут работать с секвенатором и анализировать результаты.
Планируется использовать NGS-платформы для молекулярной диагностики других заболеваний, в том числе рака, а также приобрести оборудования для синтеза олигонуклеотидов (праймеров).
На заданный вопрос докладчик ответил, что сейчас в Зимбабве нет случаев коронавирусной инфекции.
Николай Кулемин, к.б.н., ФГБУ Федеральный научно-клинический центр физико-химической медицины ФМБА России (Москва), выступил с докладом «Система анализа и хранения результатов секвенирования нового поколения».
Десятилетиями в ходе работы в лабораториях копятся разнородные NGS-данные, хочется просто ими управлять и максимально эффективно использовать. Нужно систематизировать работу во всех подразделениях и аффилированных компаниях так, чтобы максимально эффективно применять оборудование и полученные знания.
Цель — создать высокоэффективную систему управления, хранения и анализа любых NGS-данных.
Результаты NGS — это в подавляющем большинстве FASTQ.GZ файлы. Они текстовые, парные, весят 1–10 гБ (бывает ближе к 100 гБ).
При секвенировании генома или экзома человека возникают так называемые «технические повторы» — постановка одной библиотеки с несколькими различными баркодами / на различные дорожки / на разные ячейки.
Ожидаемые результаты могут быть типовыми (анализ экзомов и геномов человека, GWAS, анализ дифференциальной экспрессии) и исследовательские (разработка новых методов анализа, работа с новыми организмами).
Самая главная проблема — считать или хранить. Если хранить все промежуточные шаги анализа генома, объем будет порядка 500 гБ.
Но fastq нужно хранить по закону (США, ЕС). И вообще лучше сохранять запуски целиком на всякий случай (например, конфликта с заказчиком). И нужно хранить VCF/gVCF, потому что с ними работают врачи.
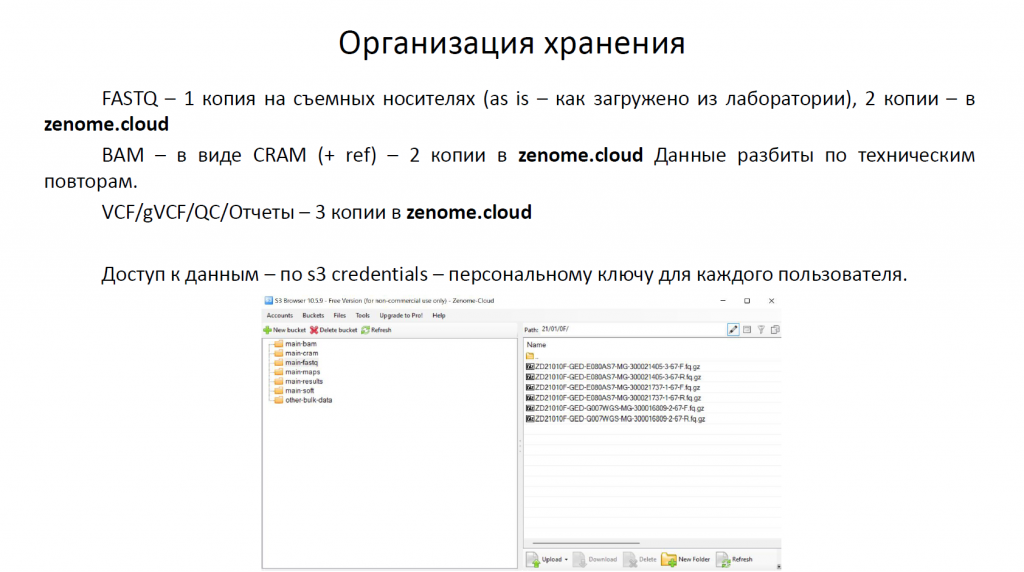
Хранить можно на переносных носителях, RAID-структурах, в облачных хранилищах. Докладчик и его команда использовали последний вариант, а также CEPH — открытую систему управления облачными хранилищами (s3.zenome.cloud). Такая система не зависит от дисков, контроллеров и так далее.
Как организовано хранение: одна копия FASTQ хранится отдельно на съемных носителях (как загружено из лаборатории), две копии — в zenome.cloud. BAM — в виде CRAM, две копии в zenome.cloud. Данные разбиты по техническим повторам. VCF/gVCF/QC/Отчеты — три копии в zenome.cloud.
Преимущество — можно разводить разные группы, у которых должен быть доступ к разным объектам.
Еще проблема — теряются описания к запускам. Поэтому придумали систему наименований, которая содержит всю информацию.

У каждого разработчика — собственная машина, к которой он имеет полный доступ через сеть. Одна команда — одна машина. Управление с использованием мессенджера Telegram. Все это позволило упростить работу.
Артем Ермаков, к.б.н., ФГБУН Институт теоретической и экспериментальной биофизики РАН (Пущино), представил доклад «Российский нанопоровый секвенатор и развитие отечественного нанопорового секвенирования». Сначала он рассказал, почему так важно заниматься нанопоровыми секвенаторами, а также обозначил достижения нанопорового секвенирования от Oxford Nanopore, такие как значительное повышение точности и дуплекс-секвенирование.
Основные элементы нанопорового секвенатора — сенсор, декодирующий многоканальный ASIC чип, сопрягающее устройство, мембрана, нанопора, моторный белок (хеликаза) и адаптер, а также программное обеспечение для декодирования пикоамперных токов нанопоры в последовательность нуклеотидов и для идентификации нуклеотидных последовательностей.
Три года назад начался проект по созданию отечественного прибора для нанопорового секвенирования. Проект создавался совместно с Институтом теоретической и экспериментальной биофизики, Институтом инженерной физики и Физтехом.
Итог первого этапа программы — создание улучшенного «клона» портативного секвенатора (Нанопорус). Улучшили электронику и создали собственный корпус, позволяющий работать в широком диапазоне температур (оригинальный корпус не выдерживает температуру выше 25 градусов).
Сейчас уже подготовились к серийному выпуску этих приборов. Есть предзаказ на выпуск 50 приборов, которые будут произведены в ближайшие 4–5 месяцев. Распространитель — компания SkyGen. На раннем этапе есть возможность поставить прибор в лаборатории, заинтересованные в нем.
Прибор совместим с оригинальной химией Oxford Nanopore и программным обеспечением.
Проточные секвенирующие ячейки можно регенерировать — на этом построена работа.
Использовать фосфолипидные мембраны нельзя, потому что они не выдерживают разности потенциалов и быстро окисляются. Поэтому используются амфифильные триблоксополимеры. Технология нанесения мембран уже отработана.
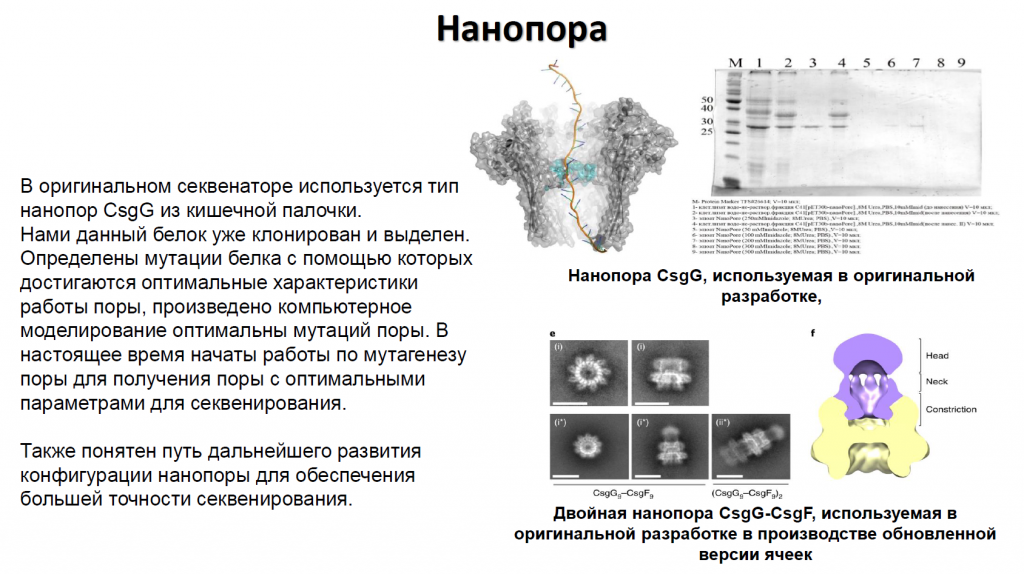
Также уже клонирован и выделен белок нанопоры, определены мутации белка, с помощью которых достигаются оптимальные характеристики работы поры, произведено компьютерное моделирование оптимальных мутаций поры. В настоящее время начата работа по мутагенезу поры. Понятен путь дальнейшего развития конфигурации нанопоры для обеспечения большей точности.
Адаптеры осуществляют проталкивание цепи нуклеотидов в нанопору. Они достаточно сложно устроены. С помощью компании «Синтол» удалось синтезировать все компоненты адаптера. Получена водорастворимая форма хеликазы. До нового года планируется испытание своего набора для пробоподготовки на основе собственного адаптера и хеликазы. Разработчики определили ключевые мутации, оптимизирующие работу хеликазы. Идут работы по ее клонированию и мутагенезу.
Программное обеспечение важно для создания мембраны, внесения нанопор на мембрану сенсора, для тестирования и контроля качества. Было модифицировано оригинальное программное обеспечение.
Разработчики собираются получить РУ, пока только на прибор. Была проведена валидация, разработка не отличается от оригинала. Планируются сличительные испытания с оптическими секвенаторами.
Алина Мацвай, к.б.н., ФГБУ «Центр стратегического планирования и управления медикобиологическими рисками здоровью» ФМБА России (Москва), выступила с докладом «Универсальный высокопроизводительный метод обнаружения широкого спектра вирусных нуклеиновых кислот для контроля циркуляции инфекционных агентов».
При превентивном мониторинге объектами исследования могут быть разные образцы —окружающей среды, смывы поверхностей (например, в местах массового скопления людей), образцы от домашних и диких животных, а также клинический материал.
Из образцов выделяют НК и анализируют несколькими молекулярно-генетическими методами, чтобы выявить НК патогенов. После этого идут более глубокие исследования с получением культур патогенов (если возможно), исследования генома.
Молекулярно-генетические методы — это довольно просто, дешево и быстро. Идеальный молекулярно-генетический метод скрининга: универсальный (подходящий и для новых патогенов), информативный, прямой (без культивирования), высокопроизводительный, достаточно дешевый.
RT-ПЦР удовлетворяет многим требованиям. Но он позволяет выявить только ограниченный спектр уже известных патогенов.
NGS позволяет не только выявить патоген, но и охарактеризовать его со всех сторон. Он подходит для выявления новых патогенов. Но есть проблема с ценой.
Есть еще проблема метагеномных исследований. При их проведении мы вынуждены секвенировать множество лишних данных, таких как ДНК хозяина и его микробиома, рРНК. В среднем нагрузка НК патогена — десятые и сотые доли процента. Это делает анализ более дорогим и затрудняет его. Нельзя заранее знать, какую глубину секвенирования или мощность платформы нужно выделить на образец, чтобы получить заданную чувствительность. То есть невозможно заранее оценить чувствительность исследования. И это зависит от образца.
Применяется метабаркодирование ДНК — это таргетное обогащения, предполагает амплификацию с «штрих-кодирующими» праймерами. Эти праймеры выбираются на специальную маркерную область, которая есть у широкого круга патогенов. Но, например, для вирусов такого универсального локуса нет — вирусы слишком разные.
Может, поможет набор маркерных областей? Исследовательская группа докладчицы предложила решение. Это алгоритмический подход к анализу вирусных возбудителей, компьютерная программа, которая осуществляет автоматический выбор минимального набора наиболее консервативных областей. Областей, к которым можно подобрать паймер. И эти праймеры должны работать в режиме мультиплексной ПЦР.
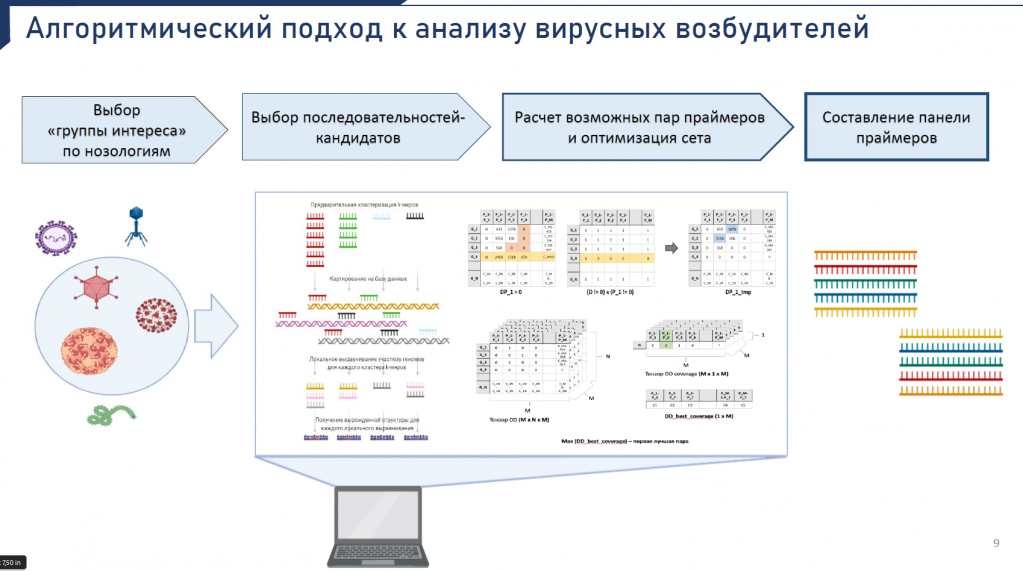
Вирусы разбивают на так называемые «группы интереса», например, по нозологиям.
Изначально берется база данных, в которую могут входить полные геномы, полные гены или любые гомологичные последовательности. На выходе получаем готовый к заказу набор праймеров и параметров к ним.
Есть два режима — быстрый и режим поиска гомологов (глубокий анализ, который позволяет в том числе выявлять новые вирусы).
Докладчица привела в пример несколько панелей, в том числе для анализа респираторных инфекций, острых кишечных инфекций и нейроинфекций. Также есть панели для скрининга патогенов птиц и рукокрылых.
Время работы программы — несколько часов. Метод подходит для патогенов любой природы, обнаружения новых вирусов, для секвенирования на любой платформе второго поколения и нанопорового секвенирования. Метод является информативным, прямым и высокопроизводительным (экономическая выгода по сравнению с метагеномикой — более чем в 40 раз).
При этом это не замена ПЦР или метагеномике. Метод должен применяться, когда мощности ПЦР-исследования уже недостаточно, а применение метагеномики еще не оправдано.
Анастасия Кадыкова, ФГБУ Федеральный научно-клинический центр спортивной медицины ФМБА России (Москва), выступила с докладом «Возможности персонифицированного медицинского сопровождения профессиональных спортсменов с учетом данных высокопроизводительного секвенирования».
Самый известные «спортивные» гены — ACE(I/D) и ACTN3.
В настоящее время известно около 200 молекулярно-генетических маркеров, ассоциированных со спортивным фенотипом и успехом. Однако эти маркеры невозможно использовать для поиска людей, талантливых в спорте. Почему?
Множество факторов определяют спортивную успешность. Физические качества — сложный для изучения комплекс признаков, имеющий полигенный характер наследования. Единичные маркеры объясняют менее 2–3% признака. Для оценки развития спортивных качеств необходим анализ множества генов и их комбинаций.
Спортивные генетики заявляют, что выявление спортивных талантов должно базироваться на верховенстве фенотипа. В настоящее время нет научных оснований для использования генетического тестирования для улучшения спортивных результатов, спортивного отбора и идентификации талантов.
Парадигма сменилась, от создания «генетического паспорта» идеального спортсмена до диагностики заболеваний на донозологическом этапе, которые возникают вследствие интенсивных физических нагрузок.
В таком случае проводится секвенирование и углубленное медицинское исследование, результаты вносятся в единую электронную медицинскую систему.
Докладчица привела результаты исследования генетической причины внезапной сердечной смерти у молодых спортсменов. Анализировался аутопсийный материал молодых людей, умерших во время интенсивной физической нагрузки, а также кровь действующих атлетов без патологических изменений сердечно-сосудистой системы.
У одного из спортсменов был найден вероятно-патогенный вариант в гене TRPM4.
В более 50% случаев не удалось найти молекулярную причину внезапной сердечной смерти.
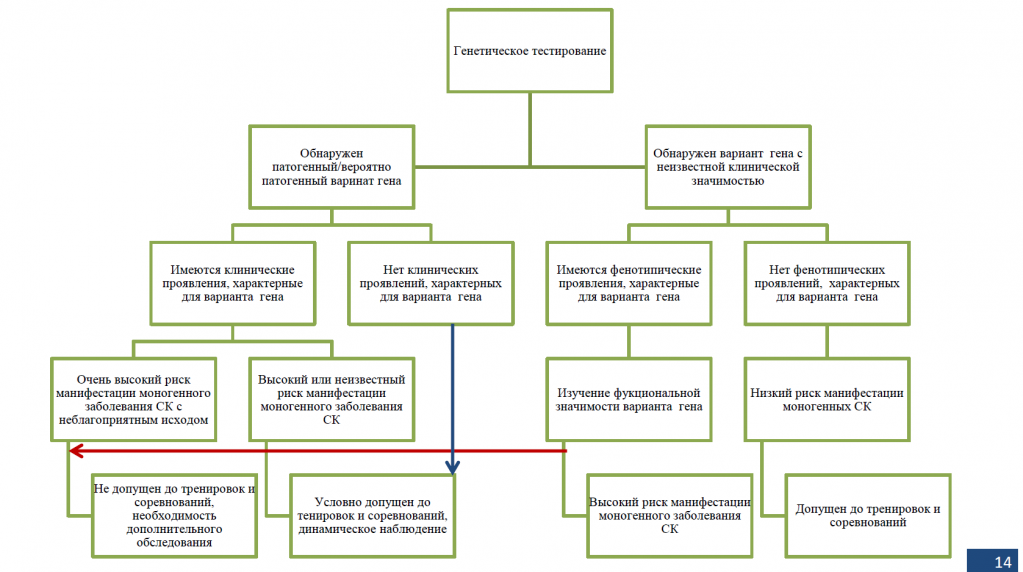
Что делать со спортсменом с вероятно-патогенным вариантом без фенотипических проявлений? На настоящий момент нет протокола. Предлагается следующий алгоритм (см. слайд). Спортсмен был условно допущен до тренировок и соревнований с динамическим наблюдением.
Оксана Рыжкова, к.м.н., ФГБНУ «Медико-генетический научный центр им. академика Н.П. Бочкова» (Москва), представила доклад «NGS: взгляд лабораторного генетика».
Есть два способа создания библиотеки — гибридизационный и амплификационный. При гибридизационном создании библиотеки видны фрагменты с разными началом и концом, то есть можно убрать дубликаты праймеров и избежать ошибки. В амплификационной системе так сделать нельзя. Вторая проблема амплификационной системы — это «drop out»-аллели.
Также качество секвенирования зависит от платформы. Кроме гомополимерных ошибок есть еще ошибки картирования.
Докладчица привела сравнительные характеристики экзомного секвенирования на разных платформах.
При переходе к геномному секвенированию ожидали, что теперь мы будем сразу получать больше клинических данных, но нет. Сейчас секвенирование генома не сильно эффективнее секвенирования экзома.
Проблема секвенирования генома — глубина прочтения. В некоторых регионах она значительно падает. В части случаев глубина покрытия не позволяет выявлять варианты, они отфильтровываются как ошибка.
Также гомологичные области генома трудно прочесть при коротких прочтениях. Еще одна проблема — CNV (copy number variation).
Докладчица делает вывод, что NGS-анализ многое дает клинике, но каждый анализ имеет свои ограничения.
Светлана Бозрова из компании «Сесана» (Москва) выступила с докладом «Новые предложения от компании “Сесана”».
Компания поставляет оборудование для полного цикла научных исследований в области молекулярной биологии, производит высококачественные реагенты для подготовки библиотек, очистки и оценки количества ДНК, предлагает профессиональную поддержку, обучение и консультацию.
Докладчица представила три основных продукта — секвенатор Genolab M, секвенатор FASTASeq 300, автоматическая станция Opentrons OT-2.
FASTASeq 300 — высокоскоростной секвенатор. Совместим с основными типами подготовки библиотек, есть функция автоматической промывки, возможность ручной загрузки библиотек. Низкая стоимость запуска. Применим для таргетного секвенирования, полноэкзомного секвенирования и метагеномных исследований.
«Сесана» производит собственные реагенты Raissol Bio. Они производятся в России. В этом году выпустили новые компетентные клетки, набор для выделения плазмид и для обработки поверхностей.
У «Сесаны» есть собственная демо-лаборатория, которая находится в Москве. Можно посетить, посмотреть, поставить собственные образцы.
Информация о докладчиках
Tonny Savadye Deckster, PhD, из National Biotechnology Authority (NBA)
Николай Александрович Кулемин, к.б.н., ФГБУ Федеральный научно-клинический центр физико-химической медицины ФМБА России (Москва)
Артем Михайлович Ермаков, к.б.н., ФГБУН Институт теоретической и экспериментальной биофизики РАН (Пущино)
Алина Дмитриевна Мацвай, к.б.н., ФГБУ «Центр стратегического планирования и управления медикобиологическими рисками здоровью» ФМБА России (Москва)
Анастасия Игоревна Кадыкова, ФГБУ Федеральный научно-клинический центр спортивной медицины ФМБА России (Москва)
Оксана Петровна Рыжкова, к.м.н., ФГБНУ «Медико-генетический научный центр им. академика Н.П. Бочкова» (Москва)
Светлана Викторовна Бозрова, «Сесана» (Москва)


 Меню
Меню





 Все темы
Все темы




 0
0