Метод smol-seq использует ДНК-аптамеры для количественной метаболомики
Основная трудность метаболомики связана с огромным разнообразием молекул, которые приходится анализировать. Чтобы повысить точность и скорость количественного определения метаболитов, канадские ученые разработали метод smol-seq — в нем метаболиты детектируются олигонуклеотидными сенсорами, которые при связывании с мишенью высвобождают ДНК-баркод. Благодаря этому количественный анализ метаболитов «превращается» в секвенирование ДНК. Авторы показали, что подход обладает высокой специфичностью и может быть мультиплексирован.

Метаболиты служат важными индикаторами состояния здоровья или работы клеток, а изменения в их уровнях могут свидетельствовать о развитии заболеваний, таких как диабет и фенилкетонурия. Однако биохимическое разнообразие метаболитов огромно, поэтому их точное и быстрое количественное определение в различных образцах, таких как ткани, плазма или отдельные клетки, — сложная задача.
«До сих пор золотым стандартом для измерения уровня метаболитов была масс-спектрометрия, но она не так доступна и не так быстра, как анализ последовательности ДНК. Мы хотели разработать метод обнаружения метаболитов с помощью секвенирования ДНК, чтобы использовать эту невероятную мощность секвенирования», — рассказала доктор Джун Тан, первый автор опубликованного в Nature Biotechnology исследования.
Ученые из Университета Торонто (Канада) разработали метод определения метаболитов с помощью ДНК-аптамеров — технологию назвали smol-seq, что можно расшифровать как «секвенирование малых молекул». В ее основе лежит способность аптамеров специфично и с высоким сродством связывать мишени. Уже сейчас известно множество аптамеров со специфичностью к разнообразным мишеням — метаболитам, флуоресцентным красителям, лекарственным средствам, токсинам.
Принцип метода smol-seq основан на использовании особого класса аптамеров, изменяющих свою конформацию при связывании с лигандом (structure-switching aptamers, SSA). Эти изменения позволяют регистрировать связывание лиганда, чаще всего через флуоресценцию или изменение проводимости.
Ученые адаптировали базовый дизайн SSA, включающий два олигонуклеотида: длинный олигонуклеотид для связывания с лигандом (LBO) и короткий высвобождаемый олигонуклеотид (SRO). При связывании лиганда с LBO происходит конформационное изменение, которое выталкивает SRO, — именно по нему можно зафиксировать взаимодействие. Каждый SSA предназначен для своей мишени и имеет уникальный ДНК-баркод — например, SSA, связывающий глюкозу, высвобождает SRO с уникальным баркодом для глюкозы. Это позволяет детектировать мишень по результатам секвенирования баркода.
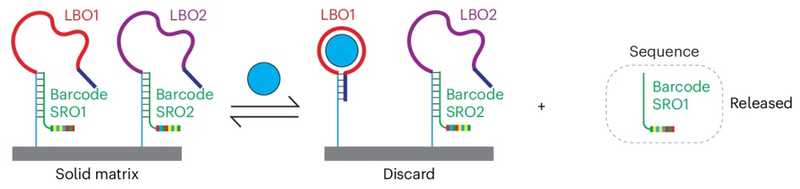 Принцип работы smol-seq: закрепленный на подложке SSA связывает мишень, благодаря чему из него высвобождается фрагмент с баркодом, который можно секвенировать.
Принцип работы smol-seq: закрепленный на подложке SSA связывает мишень, благодаря чему из него высвобождается фрагмент с баркодом, который можно секвенировать.Credit:
Nature Biotechnology (2025). DOI: 10.1038/s41587-025-02554-7 | CC BY-NC-ND
Для проверки способности баркодированных SSA обнаруживать различные мишени исследователи адаптировали существующие аптамеры или коммерческие SSA, отобранные с помощью DeepSeq. Они подтвердили возможность оценить количество мишени — при увеличении уровня лиганда происходило более интенсивное высвобождение баркодов SRO. Проверка специфичности показала, что SSA очень избирательны, например, АТФ-связывающий аптамер не реагирует на другие нуклеозидтрифосфаты, а распознающий глюкозу отличает ее от галактозы.
В реальных условиях баркодированные SSA должны быть способны точно измерять концентрации мишеней в сложных смесях, например, в клеточных лизатах или плазме. Авторы работы подтвердили такую возможность — они детектировали с помощью SSA уровень противомалярийного препарата пиперахина в клеточных лизатах и бактериальных средах и специфично измеряли количество кортизола, добавленного к дрожжевому экстракту. SSA могут использоваться для измерения не только экзогенных, но и эндогенных мишеней, например, уровня АТФ в различных штаммах Escherichia coli.
Чтобы применять smol-seq для анализа множества мишеней одновременно, его разработчики применили и проверили мультиплексирование, где каждое вещество распознается отдельным аптамерным сенсором с уникальным баркодом. Также были созданы сенсоры с различными динамическими диапазонами для повышения точности измерений.
Таким образом, smol-seq представляет собой метод, который позволяет с помощью секвенирования ДНК количественно определять метаболиты. Система баркодов в smol-seq легко интегрируется с другими методами, такими как секвенирование РНК, поэтому подход не только упрощает исследование метаболомики, но и расширяет возможности мультиомиксного анализа.
Источник
Tan, J.H., Fraser, A.G. Quantifying metabolites using structure-switching aptamers coupled to DNA sequencing. // Nat Biotechnol (2025). Published: 04 February 2025. DOI: 10.1038/s41587-025-02554-7
Цитата по пресс-релизу


 Меню
Меню





 Все темы
Все темы



 0
0