Наношарики ДНК выявят патогены в образце
Недавняя публикация в Science Advances посвящена новому методу обнаружения патогенов. Подход основан на петлевой изотермической амплификации. Продукты реакции самостоятельно собирались в наношарики, которые затем детектировались на микрофлюидном чипе. Авторы статьи продемонстрировали высокую чувствительность (до 10 копий нуклеиновой кислоты) и адаптируемость метода — с его помощью детектировали разные вирусные и бактериальные патогены, в том числе в клинических образцах.
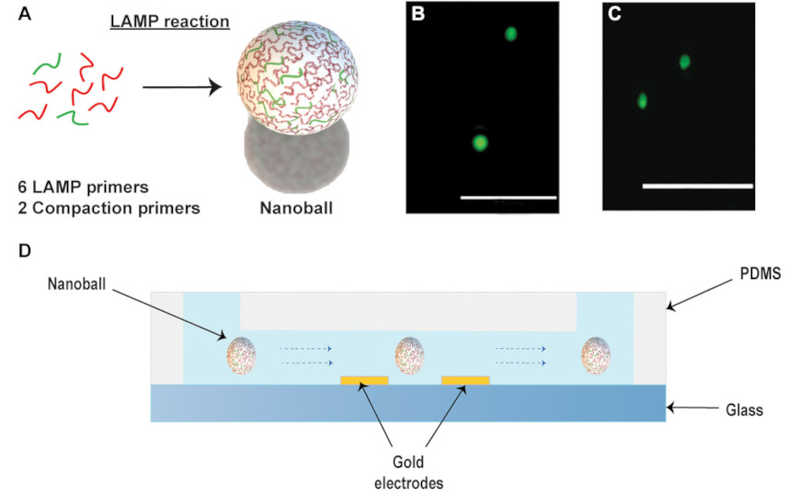
Схема работы нового подхода: продукты петлевой изотермической амплификации самостоятельно собираются в наношарики (А), изображенные на рис. B (для сравнения — наношарик диаметром 1 мкм, C). Наношарики ДНК детектируются с помощью микрофлюидного чипа (D).
Credit:
Science Advances (2023). DOI:
10.1126/sciadv.adi4997 |
CC BY
Быстрая и точная детекция нуклеиновых кислот крайне важна для идентификации патогенов. Методы обнаружения ДНК обычно подразумевают использование цветной или флуоресцентной метки, и безметочные подходы снизили бы стоимость и сложность тестов. Авторы статьи в Science Advances представили новый метод, основанный на генерации наношариков ДНК и их детектировании с помощью электрического импеданса.
Ученые модифицировали петлевую изотермическую амплификацию (LAMP-амплификацию) таким образом, чтобы в ходе реакции формировались наношарики из ДНК. Они исходили из того, что при стандартной LAMP-амплификации образуется серия конкатенированных продуктов различной длины. Авторы предположили, что можно использовать олигонуклеотиды, комплементарные общему участку, присутствующему в ампликонах, для соединения их в трехмерную структуру — наношарики. Кроме того, эти олигонуклеотиды могут уже присутствовать в реакции LAMP, что позволит генерировать наношарики ДНК параллельно с изотермической амплификацией.
В предложенной исследователями системе реакция требует шесть стандартных LAMP-праймеров, подходящих к целевой последовательности, и два дополнительных олигонуклеотида для компактизации структуры. Новый подход протестировали, применив его для выявления РНК SARS-CoV-2. Саму LAMP-амплификацию при этом дополнили этапом обратной транскрипции. Ученым удалось получить самособирающиеся компактные ДНК-наношарики диаметром от 1 до 2 мкм, которые можно было наблюдать под флуоресцентным микроскопом. Затем авторы проверили, возможно ли обнаружить эти ДНК-наношарики методом импедансной детекции в специальном микрофлюидном устройстве.
Микрофлюидный чип, который использовали ученые, был изготовлен из полидиметилсилоксана на стеклянной подложке и содержал золотые электроды. Ток жидкости в этом чипе пассивен и обеспечивается капиллярным эффектом — это исключает необходимость использования сложных насосных систем. Наношарики ДНК в таком потоке перемещаются по каналу над золотыми электродами, и когда такой шарик проходит мимо электрода, он изменяет электрическое поле между двумя электродами. Из-за этого импеданс системы меняется, и на выходе считывающей системы появляется пик сигнала.
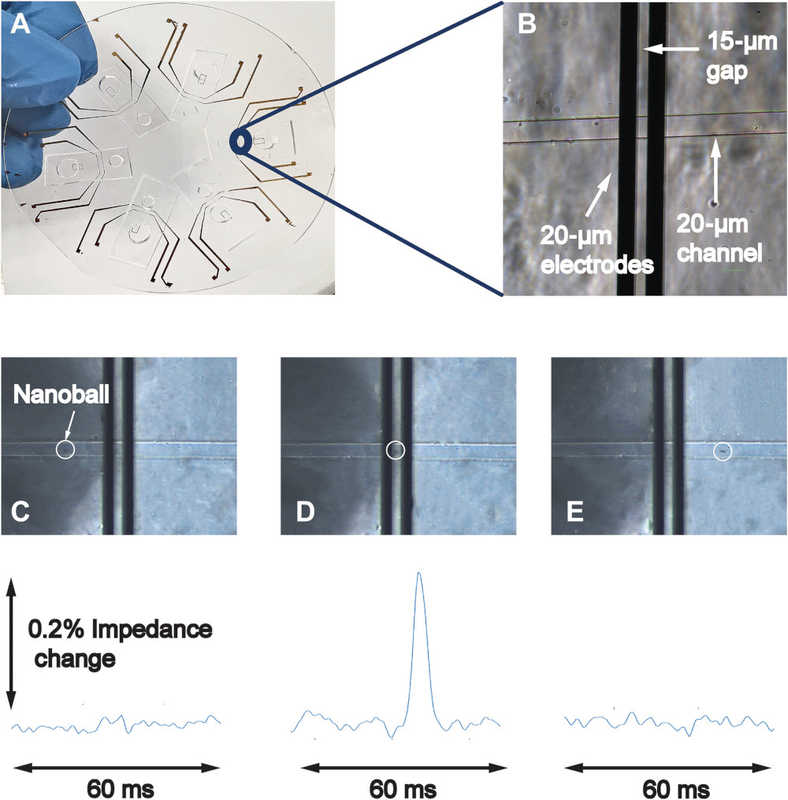
Принцип детекции: прохождение наношарика ДНК мимо золотых электродов временно изменяет импеданс всей системы, и это изменение регистрируется считывающей системой.
Credit:
Science Advances (2023). DOI:
10.1126/sciadv.adi4997 | CC BY
Авторы использовали разные концентрации компактизующих олигонуклеотидов по отношению к праймерам (соотношение 1:1 или 9:1). Второй вариант (9:1) дал большее среднее количество детектируемых наношариков ДНК (>100 за 10 мин), поэтому ученые остановились на этом соотношении для дальнейших экспериментов.
Дальнейшая оптимизация метода затрагивала чувствительность на уровне параметров электрической схемы (например, подбор оптимального напряжения), а также структуру самих компактизующих олигонуклеотидов. После оптимизации ученые определили предел обнаружения разработанной ими методики. Оказалось, что даже самая низкая использованная концентрация синтетической РНК (10 копий РНК) была достоверно отличима от отрицательного контроля.
Чтобы проверить применимость подхода, авторы работы использовали его для выявления SARS-COV-2 в полученных от пациентов образцах мазков из верхних дыхательных путей. В анализ также включили пять образцов, которые по результатам RT-qPCR были отрицательны на COVID-19. Новый метод позволил исследователям выявить клинически значимые уровни вируса с помощью импедансного детектора. В соответствии с уже полученными результатами, вирусная нагрузка в образцах сильно коррелировала с количеством детектированных наношариков ДНК.
Кроме того, авторы протестировали новый метод для детекции различных клинических патогенов: вируса гриппа А/H1N1, ВИЧ и M. tuberculosis. Также они использовали праймеры, специфичные к последовательности гена β-лактамазы — распространенного гена устойчивости к антибиотикам. Подход оказался успешен во всех случаях и позволил выявить целевые патогены в образцах. Это, по словам ученых, наглядно демонстрирует, что любую LAMP-амплификацию можно достаточно легко адаптировать к новой стратегии к стратегии «LAMP-компактизации» в сочетании с импедансной детекцией.
Таким образом, ученые продемонстрировали пассивную проточную импедансную детекцию новых безметочных ДНК-наношариков, нацеленных на SARS-CoV-2, а затем на различные патогены вирусного или бактериального происхождения. Новый подход, по словам авторов, потенциально позволяет создать чувствительную (10 копий), дешевую, быструю (менее 60 минут) и масштабируемую систему детекции патогенов, которую можно будет применять у постели больного.
Источник
Muhammad Tayyab et al. Digital assay for rapid electronic quantification of clinical pathogens using DNA nanoballs // Sci Adv (2023). DOI: 10.1126/sciadv.adi4997


 Меню
Меню





 Все темы
Все темы



 0
0