Новая платформа для высокопроизводительной детекции коронавируса
Канадские ученые описали в Nature Communications процесс разработки и наладки платформы C19-SPAR-Seq. Платформа совмещает мультиплексную ОТ-ПЦР с NGS и предназначена для скрининга популяции на COVID-19.
Credit: Marie-Ming Aynaud, et al. (2021); DOI: 10.1038/s41467-021-21653-y | CC BY 4.0
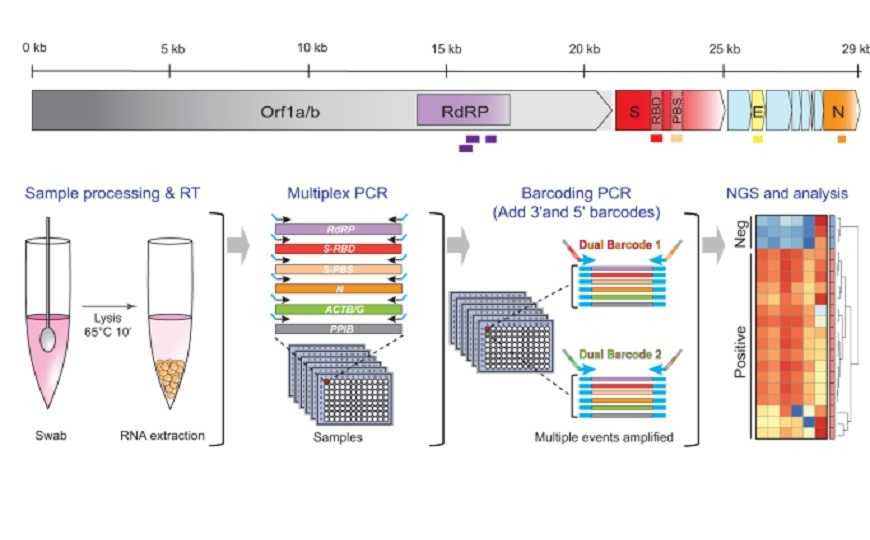
Канадские ученые разработали мультиплексную автоматизированную платформу для детекции РНК SARS-CoV-2 в клинических образцах с одновременным секвенированием амплифицированных последовательностей. Платформа получила название C19-SPAR-Seq (Systematic Parallel Analysis of RNA coupled to Sequencing for Covid-19 screening).
Система основана на NGS-технологии SPAR-Seq, разработанной этой же командой ранее. На первом этапе проводится обратная транскрипция, а затем ПЦР для выявления РНК SARS-CoV-2 в образце с использованием мультиплексной смеси праймеров. В качестве мишеней изначально были выбраны гены РНК-зависимой РНК-полимеразы (RdRP), нуклеокапсида (N), белка оболочки (E), два сайта гена S-белка, соответствующие рецептор-связывающему домену (Srbd) и многоосновному сайту расщепления (Spbs), а также праймер к человеческому гену в качестве контроля. Последние две мишени захватывают последовательности, специфичные именно для SARS-CoV-2, тогда как праймер к RdRP сработает и в случае вируса SARS-CoV, распознать который можно с помощью секвенирования.
После амплификации на 5’- и 3’-концы полученных фрагментов ДНК навешиваются штрихкоды, а затем проводится секвенирование и анализ результатов.
Первичную проверку и доработку платформы C19-SPAR-Seq проводили на архивных образцах, хранящихся в больнице Маунт-Синай сети здравоохранения Университета Торонто. Для того, чтобы анализ удовлетворял строгим критериям клинической диагностики, авторы при каждом запуске использовали большое количество контрольных образцов, а также внедрили в систему алгоритм, назначающий пороговые значения для каждого цикла и оценивающий качество выходных данных. В процессе валидации на пилотной выборке из 89 коронавирус-положительных и 289 коронавирус-отрицательных образцов авторы обнаружили, что увеличение количества отрицательных образцов создает шумы в результатах C19-SPAR-Seq из-за большого количества неспецифических продуктов амплификации.
Ученые оптимизировали набор праймеров: изменили таргетную область RdRP и убрали праймеры к N и E. С этим наборов они протестировали C19-SPAR-Seq на расширенной выборке из 663 архивных образцов (назофарингеальных мазков и бронхоальвеолярного лаважа), для 98 из которых ранее было подтверждено наличие РНК SARS-CoV-2. Распределение ридов для положительных и отрицательных образцов четко различалось, при этом внутренняя система контроля C19-SPAR-Seq посчитала результат для 121 образца незначимым. В основном это были образцы БАЛ. Ученые заключили, что образцы БАЛ сильно деградируют со временем и что внутренняя система контроля C19-SPAR-Seq хорошо определяет образцы ненадлежащего качества.
В целом чувствительность и специфичность C19-SPAR-Seq составили 87% и 100% соответственно; для образцов с высокой вирусной нагрузкой — >95% и 100%. По словам авторов, платформу можно масштабировать до десятков тысяч образцов на один запуск.
«Она исключительно надежна и адаптируема, — говорит один из авторов работы Хавьер Эрнандес, младший исследователь в Институте Люненфельда-Таненбаумов (Торонто). — Цикл занимает около 24 часов. Процесс очень прост, и мы автоматизировали практически каждый его шаг».
Ученые считают, что в перспективе C19-SPAR-Seq может применяться для крупномасштабного мониторинга SARS-CoV-2 и других патогенов.
Источник
Marie-Ming Aynaud, et al. // A multiplexed, next generation sequencing platform for high-throughput detection of SARS-CoV-2. // Nature Communications 12, 1405 (2021); DOI: 10.1038/s41467-021-21653-y
Цитата по пресс-релизу


 Меню
Меню





 Все темы
Все темы



 0
0