Секвенирование Oxford Nanopore диагностирует инфекцию в протезах суставов
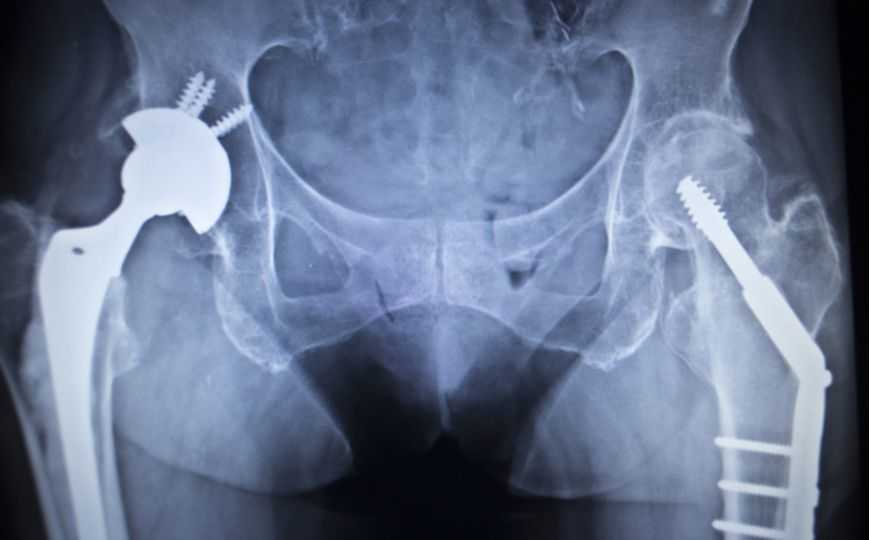
Исследователи из Великобритании разрабатывают метод диагностики инфекций в имплантированных протезах суставов, который позволит получить результаты в течение нескольких часов. Сейчас подобный анализ занимает недели или более.
О том, может ли нанопоровое секвенирование заменить культивирование при диагностике инфекций протеза, рассказала на Европейском конгрессе клинической микробиологии и инфекционных заболеваний в Амстердаме Тереза Стрит, сотрудник факультета медицины Наффилда и преподаватель генетики Института гуманитарных наук Оксфордского университета.
Чтобы проиллюстрировать проблему, Стрит привела статистические данные по Великобритании. В частности, из примерно 113 000 операций по замене коленного сустава, проведенных в 2017 году, 6500, или около 6%, были сделаны по поводу осложнений с протезом, и почти четверть этих операций были назначены из-за инфекции или подозрения на инфекцию. Что касается диагностики, золотым стандартом сейчас является бактериальная культура из образцов перипротезной ткани, собранных во время операции; другой вариант — получение культуры из обработанных ультразвуком протезов в физиологическом растворе. Однако частота обнаружения инфекционного агента в культуре из образцов ткани — всего около 65%, и процедура эта весьма сложная.
Подобную задачу может решить метагеномное секвенирование, то есть секвенирование тотальной ДНК или РНК из клинического образца и сравнение полученных последовательностей с эталонными геномами патогенов. Такую работу Стрит и ее коллеги опубликовали в Journal of Clinical Microbiology, причем использовали секвенирование Illumina MiSeq. Удаленный при операции протез помещали в физиологический раствор и обрабатывали ультразвуком, чтобы разрушить бактериальную биопленку и увеличить количество доступных клеток. Затем из образцов выделяли ДНК.
Чувствительность и специфичность достигли около 88%, но мешала ДНК человека: в 97% выборок более 90% данных относились к человеческому геному. Другой проблемой были загрязнения, возникающие при сборе и обработке образцов; приходилось генерировать для сравнения метагеномные профили образцов, не содержащих культуры.
Поскольку для более точной идентификации требовалось больше чтений, группа переключилась на секвенирование Oxford Nanopore (платформа MinIon), которое обеспечивает более длинные риды и большее покрытие эталонных геномов, и что особенно важно, поставляет данные в режиме реального времени, практически во время операции. Исследователи внесли некоторые изменения в протокол нанопорового секвенирования, уделив особое внимание снижению количества человеческой ДНК. Методы анализа последовательностей описаны в публикации 2018 года.
В неопубликованном исследовании команда Стрит проанализировала образцы ДНК, оставшиеся от секвенирования с помощью Illumina MiSeq, и получила отличное соответствие. В некоторых случаях удалось определить виды микроорганизмов, в предыдущем исследовании идентифицированные с точностью до рода (например, Bacillus cereus и B. thuringiensis вместо Bacillus spp.), причем результат был получен быстрее.
Исследователи думают о дальнейшей оптимизации метода, в частности, о том, чтобы разрушать человеческие клетки детергентом, а затем обрабатывать образец нуклеазой, чтобы удалить человеческую ДНК, и только затем лизировать бактериальные клетки. Они также тестируют набор Oxford Nanopore для быстрого ПЦР-баркодирования, который позволяет работать с такими малыми количествами, как 10 нг ДНК (а после удаления человеческой ДНК материала в образце останется немного). Этот набор также предусматривает шестиминутную стадию ПЦР-элонгации, которая создает более длинные фрагменты для анализа, и большее количество циклов ПЦР, что увеличивает выход. Кроме того, использование этого набора делает возможным мультиплексирование (объединение образцов для дальнейшей совместной обработки), что снижает стоимость секвенирования.
По словам Стрит, их данные хорошо согласовались с результатами, полученные методом культивирования, кроме того, они получили положительные результаты там, где культивирование давало отрицательные. В отдельных случаях им даже удалось идентифицировать гены устойчивости к противомикробным препаратам у стафилококка.
В одном образце исследователи обнаружили два разных вида Staphylococcus, один из которых идентифицировали как S. haemolyticus, а другой определить не смогли. Позднее с помощью масс-спектрометрии MALDI-ToF они установили, что это S. caprae. Этот вид отсутствовал в справочной базе данных, который они пользовались, и Тереза Стрит подчеркивает, что качество результатов метагеномного секвенирования зависит от базы данных в той же мере, что и от используемой технологии.


 Меню
Меню





 Все темы
Все темы



 0
0