Стала доступна обширная база геномных профилей ксенографтных опухолевых моделей
Данные геномного и транскриптомного профилирования 261 ксенографтной опухолевой модели могут упростить определение мишеней для терапии и разработку лекарственных препаратов.
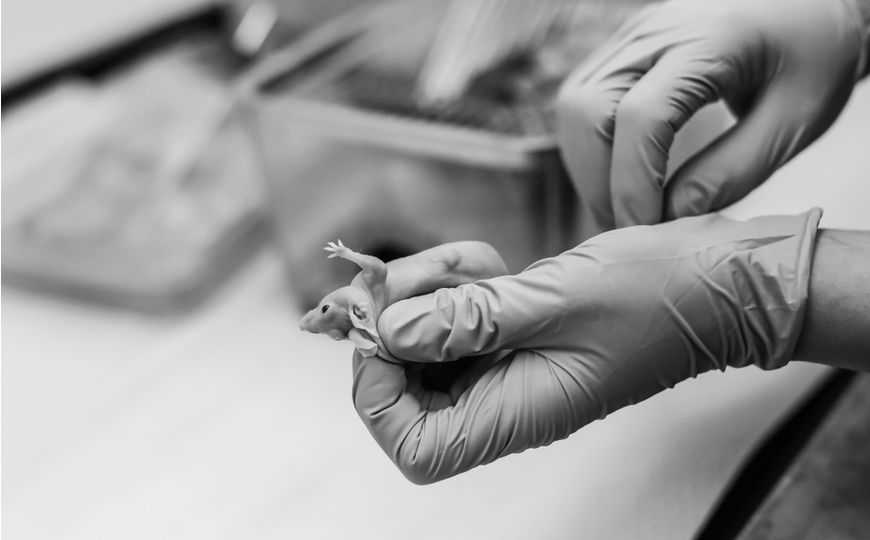
Одной из основных проблем детской онкологии является разработка быстродействующих методов лечения. Эта проблема стоит особенно остро из-за гетерогенности опухолевых тканей, различия молекулярных механизмов, отвечающих за первичное развитие опухоли и за рецидив, генетических и молекулярных отличий детских раков от взрослых, а также возрастных ограничений использования некоторых методов лечения.
В настоящее время для предсказания развития болезни и реакции на терапию широко используются животные опухолевые модели. Наиболее точной моделью считается перевивка опухоли от пациента напрямую к мыши с иммунодефицитом — ксенографтная модель (при этом раковые клетки предварительно не культивируются). В более ранних исследованиях составляли характеристики ксенографтных моделей с целью выявления оптимальных мишеней и терапевтических противоопухолевых агентов для доклинических испытаний. Однако выборки опухолей были нерепрезентативны по гистологическому разнообразию или по данным геномного анализа.
Работа, проделанная учеными из США и Австралии, включала геномный анализ 261 ксенографтной модели 29 злокачественных новообразований у детей, среди которых присутствовали различные солидные опухоли, опухоли ЦНС, гематологические и рабдоидные опухоли. Образцы собирались у пациентов во время или после прохождения курса химиорадиотерапии. Большинство образцов для создания моделей бралось сразу после постановки диагноза, но также в выборке были рецидивирующие опухоли, при этом для одного пациента составлялись пары моделей диагноз–рецидив. Для 240 моделей было проведено полноэкзомное секвенирование, для 244 — полнотранскриптомное секвенирование, у 252 моделей были проанализированы однонуклеотидные полиморфизмы и для всех моделей было проведено STR-профилирование для выявления микросателлитных последовательностей. По результатам выявлялись паттерны мутаций для разного вида опухолей и определялись тенденции в возникновении слияний генов, приводящих к возникновению онкогенных химерных продуктов. Например, для саркомы Юинга было выявлено характерное слияние генов FLI1 и EWSR1. Также были проанализированы изменения, происходящие в геноме опухоли после рецидива. В целом мутационная нагрузка для рецидивирующих опухолей была значительно выше, чем для первичных.
Помимо этого, исследователи представили алгоритм машинного обучения, который классифицирует опухоли по наличию активных или неактивных генов TP53 или NF1. Данный алгоритм еще не готов для клинических исследований, и следующим шагом должна стать проверка его предсказательной силы на ксенографтных моделях.
Собранные научной группой данные сейчас общедоступны и могут быть использованы для быстрого предсказания ответной реакции определенного типа опухолей на разрабатываемый противоопухолевый агент, что ускорит клинические испытания.
Источник
Jo Lynne Rokita, et al. // Genomic Profiling of Childhood Tumor PatientDerived Xenograft Models to Enable Rational Clinical Trial Design // Cell Reports, Vol. 29(6), P1675-1689. Nov. 05, 2019; DOI: 10.1016/j.celrep.2019.09.071


 Меню
Меню





 Все темы
Все темы



 0
0