Ультраточные риборегуляторы для диагностических систем
Ученые разработали сенсоры-риборегуляторы, распознающие однонуклеотидные мутации с высокой точностью. Новая система работает при температуре человеческого тела и может использоваться в диагностических тестах на бумажной основе.
Точечные мутации могут вызывать патологии разной степени тяжести от дальтонизма до серповидноклеточной анемии, а точечные мутации в геномах патогенов имеют большую диагностическую и клиническую значимость. Детектировать их можно на уровне транскриптов, отслеживая модификации мРНК в клетке.
Ученые из США предложили использовать для этого SNIPR — (Single-Nucleotide-Specific Programmable Riboregulators) — созданный ими новый класс риборегуляторов, специфичных к однонуклеотидным вариантам.
В природе существуют разнообразные способы регуляции трансляции. Один из механизмов использует рибопереключатель, встроенный в матричную РНК. Этот переключатель может существовать в двух конформациях. В одной из них сайт связывания рибосомы неактивен, в другой — способен связываться с рибосомой. Переход из одной конформации в другую зависит от присутствия или отсутствия лиганда переключателя. В качестве лиганда могут выступать самые разные молекулы и таким образам активировать или прекращать трансляцию этой мРНК.
Ученые уже больше десяти лет разрабатывают инженерные рибопереключатели —риборегуляторы, которые в качестве лиганда используют РНК-мишень. Такие риборегуляторы могут служить РНК-сенсорами, реагирующими на изменения РНК-мишени. Их удобство в том, что они кодируются в ДНК, осуществляют предсказуемые и программируемые межнуклеотидные взаимодействия и несут последовательность репортерного белка, с помощью которого сообщают о своем статусе.
Существующие технологии, использующие риборегуляторы in vitro, имеют ряд ограничений. Например, они могут использоваться только при высоких температурах, что несовместимо с живыми клетками, а результаты, полученные in vitro, не соответствуют реальным клеточным процессам. Авторы новой работы утверждают, что SNIPR позволяют преодолеть эти ограничения.
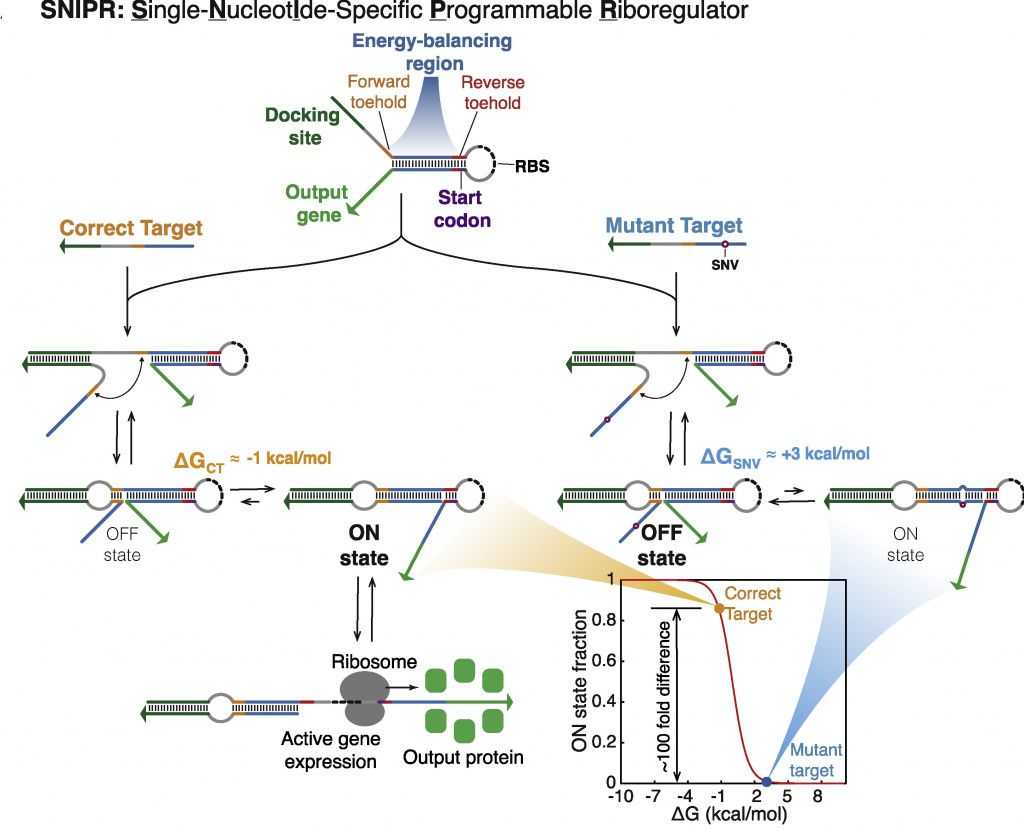
SNIPR — это молекула РНК, которая содержит коровую шпильку и состоит из четырех функциональных элементов: область балансировки энергии (energy-balancing region), стыковочный сайт, сигналы инициации трансляции и кодирующую репортерную последовательность. Область балансировки энергии — критический компонент, обеспечивающий ультраспецифичное распознавание мишени. Длина этой области определяет свободную энергию перехода между неактивным и активным состояниями. Стыковочный сайт комплементарен РНК-мишени, за счет него формируется комплекс РНК-SNIPR. До момента связывания с мишенью риборегулятор пребывает в неактивном состоянии: сайт связывания рибосомы (RBS) и старт-кодон упакованы в структуре шпильки так, что начало трансляции невозможно, репортерный белок не продуцируется. При связывании SNIPR с мишенью шпилька раскрывается, RBS и старт-кодон оказываются доступными для рибосомы, начинается трансляция репортерного белка.
Для SNIPR равновесие между активным и неактивным состояниями определяется разницей их свободной энергии. Ученые создали биохимическую модель и определили, что разница в -1 — -2 ккал/моль между неактивным и активным состояниями при идеально соответствующей мишени обеспечивает наиболее эффективную работу системы. Если хотя бы одно основание в таргетной области будет отличаться, это прибавит 4 ккал/моль и сильно сдвинет равновесие в сторону неактивного состояния.
Fan Hong, et al. По Creative Commons Attribution (CC BY 4.0)
Ученые проверили работу SNIPR in vivo в клетках Escherichia coli при 37°C. SNIPR и РНК-мишень транскрибировались в одной клетке с разных плазмид. В качестве репортерного белка выбрали GFP. Чтобы проверить, насколько специфично работает SNIPR, авторы использовали пять мутантных таргетных РНК и выяснили, что экспрессия GFP при связывании с корректной мишенью была в 100 раз выше, чем для мутантных мишеней. Дальнейшее тестирование показало, что SNIPR успешно распознает все три типах однонуклеотидных мутаций (инсерция, делеция или замена). Более того, SNIPR также пригодны для определения эпигенетических меток, что показали эксперименты in vitro на синтетических РНК, содержащих N6-метиладенозин и сайты 2′-O-метилирования.
Дизайн SNIPR разрабатывается in silico, заново для каждой мишени. Для облегчения этой задачи ученые создали компьютерный алгоритм, который формирует структуру SNIPR по входным данным: последовательности мутантной мишени, последовательности РНК дикого типа и репортерному белку. Корректность работы алгоритма проверили на мутациях ВИЧ, ассоциированных с устойчивостью к антиретровирусной терапии, а также в бесклеточных системах транскрипции-трансляции на клинически значимых раковых мутациях и других мишенях.
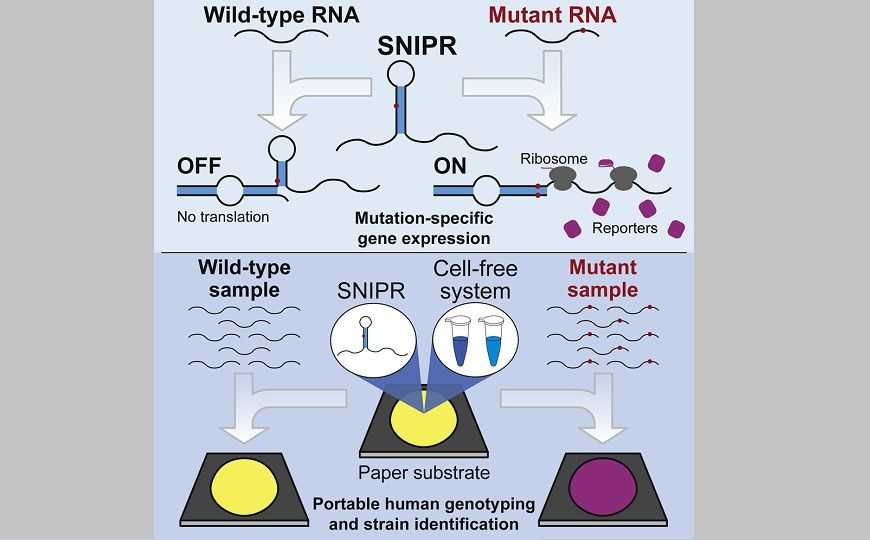
Авторы также показали возможность применения SNIPR в бесклеточных реакциях транскрипции-трансляции на бумаге. В этом случае при связывании с мишенью образуется репортерный продукт красного цвета, видимый невооруженным глазом. Компоненты реакции можно лиофилизировать на бумаге, хранить при комнатной температуре и смачивать водой перед использованием.
Мягкий температурный режим (37°C), в котором работают SNIPR, позволяет использовать технологию для недорогой диагностики в полевых условиях. Для демонстрации этого ученые использовали образцы РНК вируса Зика. Сначала они провели изотермическую рекомбиназную полимеразную амплификацию с обратной транскриптазой и праймерами, модифицированными особым образом. Затем добавили продукт к SNIPR-системе на бумажной основе и детектировали три различных штамма вируса. С небольшими дополнениями SNIPR также можно использовать для генотипирования человеческих образцов.
Таким образом, SNIPR — это высокоточный инструмент, который можно использовать для решения широкого ряда фундаментальных и прикладных задач. В частности, SNIPR имеют большой потенциал в области изучения кратковременных транскриптомных и эпитранскриптомных изменений в живых клетках и для разработки диагностических сенсоров.








 Меню
Меню





 Все темы
Все темы






 0
0