Наборы праймеров NEB обеспечивают полное покрытие генома штамма омикрон
Мутации в новых штаммах коронавируса влияют не только на точность диагностики COVID-19, но и на секвенирование SARS-CoV-2 по стандартным протоколам. Компания New England Biolabs разработала решение по протоколу ARTIC, позволяющее секвенировать омикрон с полным покрытием.
ktsdesign | 123rf.com
Самый распространенный метод секвенирования SARS-CoV-2 — ампликонное секвенирование по протоколу ARTIC. Первый этап протокола, мультиплексная ПЦР, необходим для обогащения вирусных последовательностей в клинических образцах. Однако мутации в геноме коронавируса могут повлиять на эффективность связывания праймеров с мишенями. Сообщество ARTIC Network отслеживает мутации и обновляет наборы праймеров. Последняя версия праймеров ARTIC V4, разработанная в июне 2021 года, учитывает мутации восьми линий SARS-CoV-2: B.1.1.7 (альфа), B.1.351 (бета), B.1.429 (эпсилон), B.1.525 (эта), B.1.617.1 (каппа), B.1.617.2 (дельта) и P.1 (гамма). В ноябре 2021 года впервые был выявлен штамм омикрон. Он несет большое количество уникальных мутаций, а потому есть риск, что при секвенировании по протоколу ARTIC V4 его покрытие будет недостаточным.
Производители наборов для секвенирования SARS-CoV-2 стремятся обеспечить максимальное покрытие вирусного генома. Компания New England Biolabs вывела на рынок линейку продуктов для секвенирования по протоколу ARTIC, которая позволяет с высокой эффективностью закрыть почти все регионы генома омикрона. Решение NEB включает два сета праймеров. Первый — уникальный набор VarSkip Short, разработанный специалистами NEB для уменьшения влияния новых штаммов, таких как омикрон и дельта. Второй представляет собой классический набор ARTIC V3.
Сравнение разных наборов праймеров на клиническом образце, содержащем омикрон, показало, что VarSkip Short успешно выявляет этот штамм. Выпадают только ампликоны 56 и 67, и еще для двух ампликонов, 20 и 64, покрытие снижено. При этом набор ARTIC V3 перекрывает эти области. Два сета праймеров в наборе NEB позволяют создать две библиотеки для одного образца и обеспечить таким образом 100% покрытие (рис. 1)
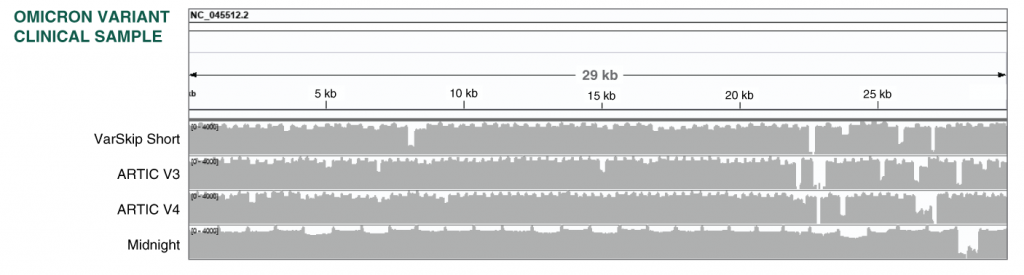 Рисунок 1. Покрытие генома SARS-CoV-2 омикрон из клинического образца при секвенирование с праймерами NEBNext VarSkip Short SARS-CoV-2, NEBNext ARTIC SARS-CoV-2 primer pools (ARTICv3), MilliporeSigma ARTICv4, IDT Midnight-1200.
Рисунок 1. Покрытие генома SARS-CoV-2 омикрон из клинического образца при секвенирование с праймерами NEBNext VarSkip Short SARS-CoV-2, NEBNext ARTIC SARS-CoV-2 primer pools (ARTICv3), MilliporeSigma ARTICv4, IDT Midnight-1200.
Решение NEB с двумя наборами праймеров доступно как для платформы Illumina, так и для Oxford Nanopore Technologies. Оно включает продукты NEBNext ARTIC SARS-CoV-2 FS c фрагментазой для подготовки библиотек (Illumina), модуль NEBNext ARTIC SARS-CoV-2 для подготовки библиотек (Oxford Nanopore Technologies) и NEBNext ARTIC SARS-CoV-2 модуль для RT-PCR.
Подробнее о продуктах NEB для секвенирования по протоколу ARTIC — на сайте компании SkyGen, дистрибьютора NEB в России.
Партнерский материал


 Меню
Меню





 Все темы
Все темы




 0
0