Нанопоровое секвенирование быстро идентифицирует вторичные инфекции у пациентов с COVID-19
Британские ученые провели нанопоровое секвенирование метагенома для выявления вторичных бактериальных и грибковых инфекций у пациентов с COVID-19, подключенных к аппарату искусственной вентиляции легких. Этот метод позволяет в день взятия пробы идентифицировать патоген и показать, есть ли у него резистентность к антибиотикам. Вспышку внутрибольничной инфекции можно расшифровать в течение суток с помощью последующего молекулярного типирования.
Применение искусственной вентиляции легких (ИВЛ) часто приводит к попаданию в легкие микроорганизмов и развитию вторичных инфекций. Это основная причина смерти пациентов на ИВЛ; особо остро эта проблема встала во время пандемии. Стероидная терапия, которую получают пациенты с COVID-19, может усугублять развитие инфекции.
Обычно пациентам отделения реанимации и интенсивной терапии (ОРИТ) с подозрением на вторичные инфекции назначают стандартную терапию, состоящую из антибиотиков или антигрибковых препаратов, до получения результатов посева. Посев занимает 2–4 дня. Назначение лечения «вслепую» и задержка постановки точного диагноза снижает эффективность лечения и создает условия для развития множественной лекарственной устойчивости у микроорганизмов. В связи с высокой загруженностью ОРИТ в пандемию встал вопрос о быстрой комплексной диагностике бактериальных и грибковых инфекций. Британские исследователи предложили внедрить в клиническую практику нанопоровое секвенирование метагенома. Этот метод позволяет охватить широкий спектр патогенов и получить результаты намного быстрее, чем посев.
Авторы использовали ранее разработанный ими алгоритм метагеномного анализа. Человеческие клетки в образце лизируют сапонином, после чего ДНК разрушают ДНКазой. Целые микроорганизмы осаждают и отмывают, после чего выделяют и секвенируют их ДНК. С помощью этого метода результаты диагностики можно получить в день взятия пробы. Дальнейший анализ позволяет выполнять молекулярное типирование микроорганизмов.
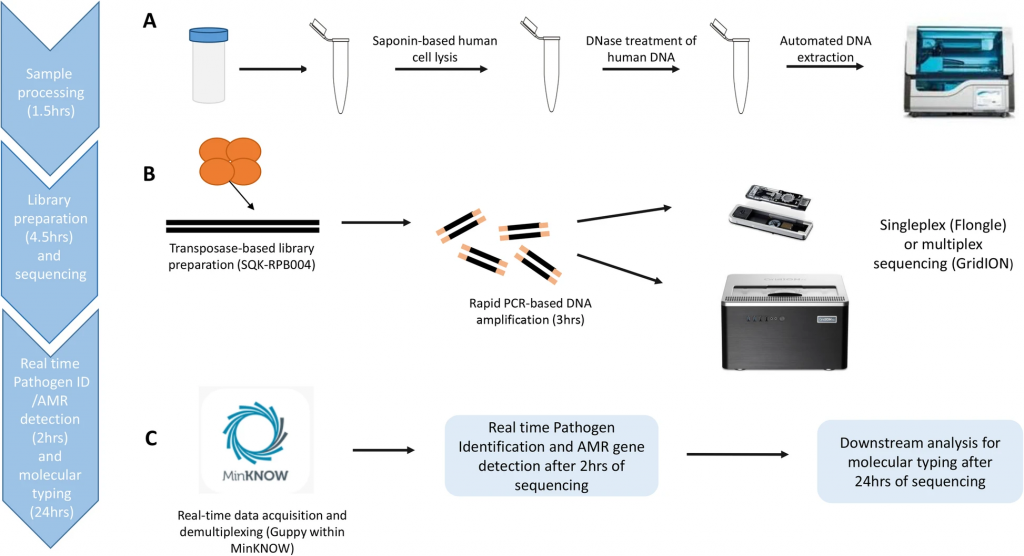 Этапы метагеномного анализа. А. Подготовка образца (1,5 часа). Лизис человеческих клеток с помощью сапонина, обработка ДНКазой, экстракция ДНК микроорганизмов. В. Создание библиотеки и секвенирование (4,5 часа). Баркодирование и нанопоровое моноплексное или мультиплексное секвенирование. С. Сбор и анализ данных в реальном времени с помощью программ
MinKNOW и
Guppy (2 часа). После идентификации микроорганизмов и генов устойчивости к антимикробным препаратам можно провести молекулярное типирование патогенов. Credit: Charalampous T., et al. DOI: 10.1186/s13073-021-00991-y
Этапы метагеномного анализа. А. Подготовка образца (1,5 часа). Лизис человеческих клеток с помощью сапонина, обработка ДНКазой, экстракция ДНК микроорганизмов. В. Создание библиотеки и секвенирование (4,5 часа). Баркодирование и нанопоровое моноплексное или мультиплексное секвенирование. С. Сбор и анализ данных в реальном времени с помощью программ
MinKNOW и
Guppy (2 часа). После идентификации микроорганизмов и генов устойчивости к антимикробным препаратам можно провести молекулярное типирование патогенов. Credit: Charalampous T., et al. DOI: 10.1186/s13073-021-00991-y
Подход опробовали в клинической практике. Для этого получили 43 образца трахеальных аспиратов и бронхиальных смывов у 34 пациентов, госпитализированных с COVID-19 в ОРИТ во время первой волны пандемии. Из всех образцов отобрали пробы для стандартного микробиологического посева и лабораторного тестирования, затем образцы анонимизировали и передали исследовательской группе для метагеномного анализа.
Результаты, полученные с помощью нанопорового секвенирования метагенома, сравнивали с результатами рутинных микробиологических тестов. Метагеномный анализ продемонстрировал чувствительность в 92% и специфичность в 82% при идентификации бактерий. Данные по заражению грибком Aspergillus fumigatus полностью совпали с результатами ПЦР-анализа. Удалось также выявить наличие или отсутствие генов устойчивости к бета-лактамным антибиотикам у бактерий порядка Enterobacterales.
Кроме того, молекулярное типирование выявило две вспышки внутрибольничных инфекций, которые были вызваны бактериями со множественной устойчивостью к антибиотикам: Klebsiella pneumoniae ST307 (у четырех пациентов) и Corynebacterium striatum (у 14 пациентов).
Профессор Джонатан Эджворт, директор Центра клинических инфекционных и диагностических исследований (Лондон) и руководитель работы, сказал: «Как только началась пандемия, наши ученые поняли, что будет полезно секвенировать геномы всех бактерий и грибков, вызывающих инфекцию, у пациентов с COVID-19, находящихся в отделении интенсивной терапии. В течение нескольких недель мы показали, что он [наш метод] может диагностировать вторичную инфекцию, таргетировать лечение антибиотиками и обнаруживать вспышки намного раньше, чем это делается сейчас –– и все это на основе одного образца. Это произведет революцию в подходе к профилактике и лечению серьезных инфекций в отделениях интенсивной терапии, и теперь мы планируем предложить его в качестве клинической услуги для пациентов с COVID-19 и гриппом этой зимой».
Источники
Charalampous T., et al. Evaluating the potential for respiratory metagenomics to improve treatment of secondary infection and detection of nosocomial transmission on expanded COVID-19 intensive care units // Genome Medicine, volume 13, article number: 182 (2021) DOI: 10.1186/s13073-021-00991-y
Цитата по пресс-релизу


 Меню
Меню





 Все темы
Все темы




 0
0