Нейронные сети помогают реконструировать структуру мозговой ткани
Привлечение машинного обучения и нейронных сетей к расшифровке изображений, полученных STED-микроскопией, повысило точность реконструкции структур нервных тканей и в сотни раз сократило время, необходимое для выполнения этой работы. Технология, названная авторами LIONESS, также позволяет изучить динамические изменения в ткани мозга.
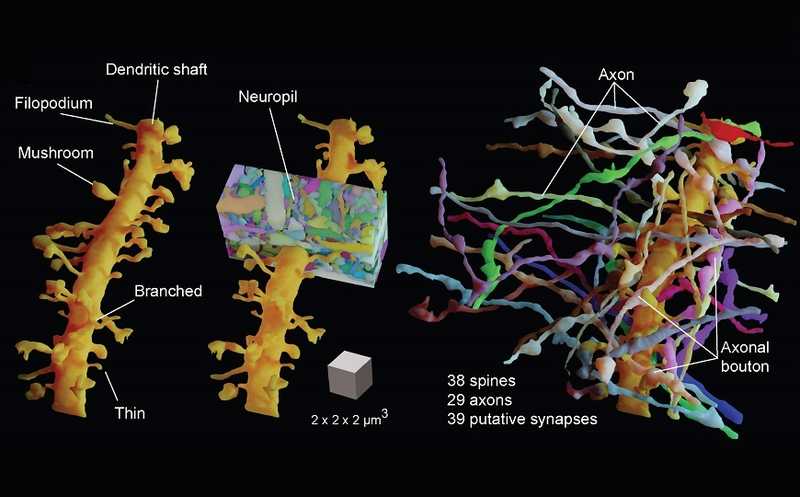
LIONESS реконструирует структуры в образце нервной ткани очень быстро и с высоким разрешением
Credit:
Johann Danzl | Пресс-релиз
Для изучения функций мозга необходим инструмент in silico реконструкции структуры нервной ткани. Электронная микроскопия реконструирует мозговую архитектуру вплоть до отдельных синаптических связей, но ограничена статичными изображениями. В то же время световая микроскопия позволяет фиксировать динамические изменения, но не обладает достаточным разрешением. Статья, опубликованная в Nature Methods, представила новый метод, объединяющий световую микроскопию и машинное обучение для получения изображений нервных тканей с высоким разрешением.
Метод представляет собой модификацию STED-микроскопии — флуоресцентной микроскопии, которая дает разрешение сверх дифракционного предела за счет избирательного подавления флуоресценции. Исследователи оптимизировали параметры STED для повышения отношения сигнала к шуму (SNR), однако результаты оставались неудовлетворительными, и они прибегли к помощи машинного обучения. Нейронная сеть была обучена на наборе спаренных STED изображений с высокой и низкой выдержкой (выдержка в данном методе напрямую влияет на разрешение и четкость изображения). На тестовом наборе данных нейронная сеть успешно реконструировала изображения, снятые с низким разрешением, без потери клеточной структуры. Это позволило ученым проводить съемку множества изображений с малым разрешением и затем реконструировать структуры в высоком разрешении при помощи нейронной сети.
Следующий шаг анализа — сегментирование (аннотация) полученных изображений, то есть выделение в них объектов и областей. Этот процесс крайне трудоемок: на сегментацию 400 нм3 ткани подготовленному ученому требуется около 450 часов. Авторы создали еще одну нейронную сеть, натренированную на небольшом (для участка размером около 285 нм3) аннотированном наборе данных. Далее сеть реконструировала больший участок ткани, который, после внесения исправлений, был использован как дополнительный тренировочный сет с общим объемом тренировочных данных около 800 нм3. Компьютерная сегментация все еще требовала верификации человеком, но тем не менее такой подход значительно снизил затраты времени — весь процесс от съёмки до получения сегментированного изображения для фрагмента ткани размером 1737 нм3 (без учета верификации человеком) занимает меньше 45 минут. В «ручном режиме» этот же объем работы потребовал бы около 860 человеко-часов. Разработанный методологический комплекс авторы назвали LIONESS (live information-optimized nanoscopy enabling saturated segmentation).
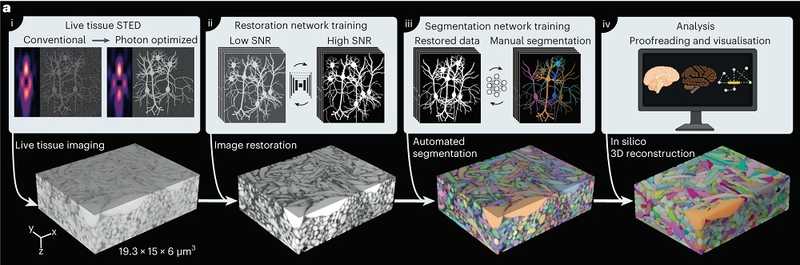 Реконструкция структуры органоида мозга человека: STED-микроскопия — обработка изображений (DL) — сегментирование (DL) — визуальный 3D-анализ. Credit: Johann Danzl |
Пресс-релиз
Реконструкция структуры органоида мозга человека: STED-микроскопия — обработка изображений (DL) — сегментирование (DL) — визуальный 3D-анализ. Credit: Johann Danzl |
Пресс-релиз
Эффективность протокола проверили на дендритных шипиках — эти структуры сложно аннотировать из-за крайне тонкой шейки, соединяющей шипик с дендритом. В качестве Однозначно идентифицировать все шипики, соединяющиеся с дендритом, можно с помощью флуоресцентных красителей, что дает объективный стандарт эффективности. Ученый, проводящий сегментацию в ручном режиме (без красителей), корректно идентифицировал 73±8,3% шипиков. Ученый, проводящий верификацию данных LIONESS, корректно идентифицировал 83±8,0% шипиков.
LIONESS также может использоваться в тандеме с другими методами. Так, его комбинация с флуоресцентными метками позволила идентифицировать на реконструированной структуре активные синаптические соединения. При этом соотнести флуоресцентную метку с конкретным синапсом удалось в 99,9% случаев, несмотря на значительно более низкое разрешение флуоресцентного изображения.
Низкая выдержка, используемая в LIONESS, дает возможность проводить серийную съемку и, как следствие, реконструировать структуру ткани в динамике. В тестах исследователи идентифицировали изменения структуры комплекса дендритов с мшистыми волокнами на промежутке в 19,5 часов.
Авторы также продемонстрировали, что LIONESS можно применять для курирования электрофизиологических экспериментов. При анализе двух нейронов, идентифицированных как связанные при помощи электрофизиологических методов, классическая световая микроскопия подтвердила их контакт, в то время как LIONESS выявила более сложную нейронную структуру, лежащую в основе связи.
Наконец, исследователи показали возможность реконструкции структур на больших объемах путем съемки перекрывающихся изображений. Для этого был реконструирован участок нескольких аксонов кумулятивной длиной примерно в 3мм.
Таким образом, LIONESS открывает новые возможности для нейробиологических исследований, позволяя реконструировать ткань мозга с недоступными ранее скоростью и качеством.
Флуоресцентная метка из ДНК с уникальным разрешением для микроскопии единичных молекул
Система AMATERAS позволяет рассмотреть миллион клеток на одном изображении
Источник
Velicky, P., et al. Dense 4D nanoscale reconstruction of living brain tissue. // Nature Methods, published July 10, 2023. DOI: 10.1038/s41592-023-01936-6


 Меню
Меню





 Все темы
Все темы




 0
0