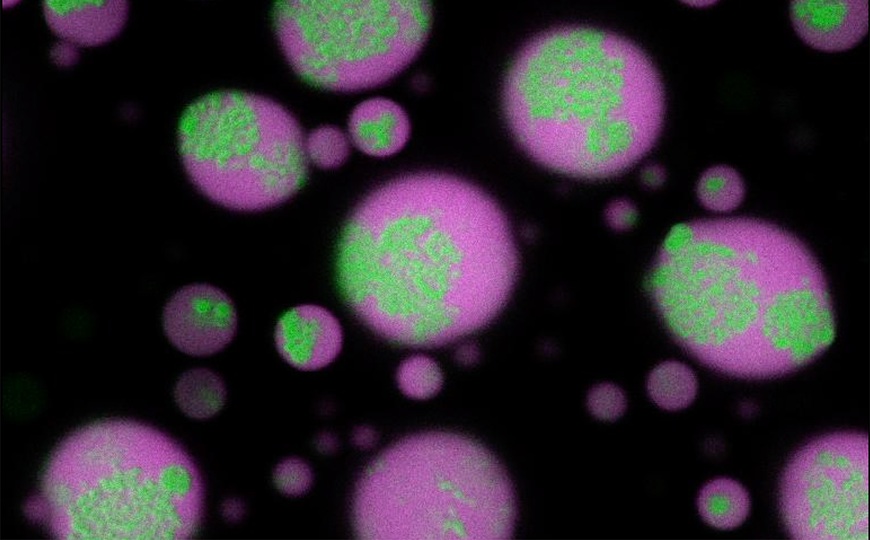
Новый метод секвенирования позволяет одновременно изучить метилом, транскриптом и доступность хроматина единичных клеток
Исследователи из США разработали метод snmCAT-seq для изучения единичных клеток. С его помощью они проанализировали роль транскрипционных факторов в дифференцировке нейронов головного мозга, а также проследили взаимодействия метилома и транскриптома. Кроме того, исследователи нашли новые подгруппы нейронов, которые связаны с биполярным расстройством, шизофренией и расстройствами аутистического спектра.
Мультиомиксный подход в секвенировании единичных клеток — один из перспективных методов для всестороннего изучения тканей и органов. Однако из-за особенностей протоколов сложно изучить одновременно несколько характеристик клеток. Ученые из США разработали способ одновременного получения данных о транскриптоме, метиломе и доступности хроматина на уровне единичных клеток — snmCAT-seq (single nuclear methylcytosine, chromatin accessibility, transcriptome sequencing). В новой методике для секвенирования используются как клетки, так и ядра. Это помогает точнее анализировать замороженные образцы, так как в ходе заморозки ядро остается более стабильным, чем клеточная мембрана.
Для разделения прочтений генома и транскриптома ученые использовали метилированный дезоксицитозинтрифосфат на стадии обратной транскрипции мРНК. После обработки образцов бисульфитом, неметилированный цитозин трансформировался в урацил. Так как в кДНК весь цитозин был метилирован, на нее эта операция никак не влияла. В то же время геном млекопитающих содержит только 5% метилированного цитозина. Следовательно, большинство цитозинов будет трансформировано в урацил, а после секвенирования геномную ДНК можно будет определить по низкой доле этого нуклеотида.
Так как динуклеотиды CpG в геноме млекопитающих метилинованы на 70–80%, прочтения с ДНК и РНК авторы разделяли на основе уровня метилирования динуклеотидов mСH, где H-любой нуклеотид, кроме гуанина. Используя этот подход, они смогли классифицировать прочтения метилома и транскриптома с точностью 99,997% и 99,97% соответственно.
Для изучения доступности хроматина исследователи использовали принцип метода NOMe-seq. В этом случае ДНК обрабатывали GpC-метилтрансферазой M.CviPI и сортировали ядра по уровню флуоресценции.
С помощью нового метода ученые нашли интересные закономерности в нейронах человека из образцов лобной доли. Они обнаружили разный уровень корреляции между метилированием mCH и уровнем экспрессии гена. Наряду с ситуацией, когда метилирование вело к ингибированию экспрессии, ученые наблюдали и обратную картину. Гены со значительной корреляцией между уровнем метилирования и экспрессии обычно были длиннее, экспрессировались на более высоком уровне и демонстрировали большую доступность хроматина. Также эти гены часто несли нейрон-специфичные функции.
Исследователи нашли гены, экспрессия которых не зависела от метилирования, а регулировалась за счет таких механизмов, как модификации гистонов.
Интегрированный метод анализа позволил выделить наиболее важные факторы транскрипции для дифференцировки нейронов. Например, на основе данных о метиломе и транскриптоме ученые предсказали вклад факторов NFIB и NFIX в спецификацию клеток каудального ганглионарного бугорка. В то же время близкородственные им факторы NFIA и NFIC не играли существенной роли в этом процессе. Более того, исследователи подтвердили существование транскрипционных факторов, для которых гиперметилирование участка связывания более предпочтительно, чем гипометилирование. До этого такие результаты демонстрировали только эксперименты in vitro.
Наконец, ученые определили типы клеток, ассоциированные с психиатрическими заболеваниями. Так, они обнаружили, что с шизофренией связано несколько типов интрателэнцефалических нейронов. Также были обнаружены новые группы нейронов, ассоциированные с расстройствами аутистического спектра и биполярным расстройством. Предыдущие исследования по поиску ассоциированных с неврологическими состояниями нейронов не сообщали о подобных результатах.
Новый метод позволил провести валидацию классификации клеток. Ранее не существовало возможности сравнить одновременно метилом и транскриптом в одной клетке. Многие исследования объединяли данные из двух разных экспериментов, поэтому валидация кластеризации клеток была невозможна. По мнению авторов, snm-CAT-seq может стать протоколом для проверки возможности такого объединения данных.
Источник
Chongyuan Luo, et al. Single nucleus multi-omics identifies human cortical cell regulatory genome diversity // Cell Genomics 2, 3 (2022), published: 9 March 2022. DOI: 10.1016/j.xgen.2022.100107


 Меню
Меню





 Все темы
Все темы




 0
0