Создана панраковая протеомная карта 949 клеточных линий человека
Исследователи из Австралии и Великобритании изучили протеом 949 клеточных линий рака, которые представляют 28 типов тканей и 40 различных онкозаболеваний. Выявлены тысячи потенциальных биомаркеров, связанных с чувствительностью рака к терапии, причем некоторые из этих биомаркеров не обнаруживались при анализе транскриптома.
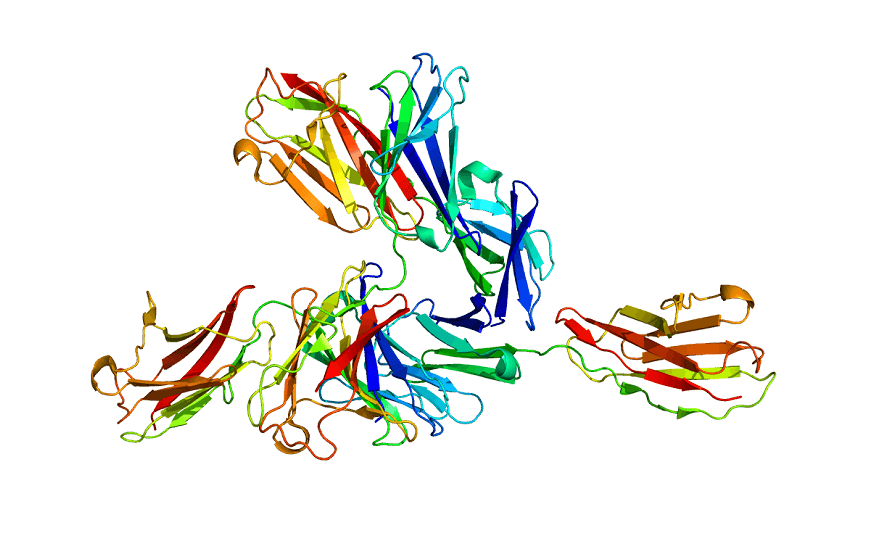
Один из значимых белков, обнаруженных авторами, — басигин (BSG)
Credit:
wikimedia.org
Анализ протеома, раскрывающий взаимосвязь генотипа и фенотипа, может повысить качество диагностики и лечения онкозаболеваний. Для того, чтобы предсказать протеом тканей раковой опухоли, недостаточно генетических и даже транскриптомных данных, поэтому необходимо изучать протеом напрямую. Масштабные исследования протеома рака затруднены из-за недостаточного количества образцов и ограничения в работе с некоторыми типами рака. Однако можно изучать протеом на клеточных линиях, которые широко использутся в качестве модели канцерогенеза.
Группа ученых из Австралии и Великобритании составила протеомную карту клеточных линий рака. В общей сложности исследователи определили 8498 белков в 949 клеточных линиях.
Первичные образцы брали из опухолей 40 генетически и гистологически различных типов рака. В качестве контроля использовали линии клеток эмбриональной почки человека HEK293T. Белки определяли методом DIA-MS (data-independent acquisition mass spectrometry). Чтобы повысить скорость обработки образцов, использовали вычислительный конвейер на основе глубокого обучения. Полученный набор данных — ProCan-DepMapSanger — в сочетании с другими фенотипическими и молекулярными данными не только идентифицировал ранее известные биомаркеры, но и помог выделить новые.
Чтобы интегрировать протеомные данные с молекулярными (метилирование промотора, экспрессия генов и количества белков) и фенотипическими (реакция на лекарство) наборами данных, доступных для большинства линий раковых клеток, авторы применили мультиомный факторный анализ MOFA. Они выявили, в частности, корреляцию между транскриптомными и протеомными данными по маркерам эпителиально-мезенхимального перехода. Известно, что эти маркеры выявляют метастазирование рака и резистентность к терапии.
Ученые отметили, что среда, в которой находились клетки, и условия роста не влияли на особенности экспрессии белков, однако размер клеток и скорость роста до некоторой степени оказались связаны с активностью ряда генов.
Авторы использовали свои данные для предсказания новых белок-белковых взаимодействий (PPI) и определили 1182 предполагаемых PPI.
Потенциальные биомаркеры ответа на терапию авторы нашли, объединив протеомные данные с результатами по чувствительности к лекарствам и данными по значимости генов, полученными с помощью CRISPR-Cas9. В этом исследовании использовали данные о 625 уникальных противораковых препаратах — на 48% больше, чем в предыдущей работе — для 947 из 949 линий раковых клеток. Эффективность 57 препаратов оказалась связана с содержанием их канонической белковой мишени или мишеней, еще для 132 препаратов были выявлены значимые ассоциации с белками, функционально связанными с их мишенями.
Авторы выявили 7698 ассоциаций лекарство-белок и 5823 ассоциаций «значимость гена-белок», указывающих на возможную уязвимость к раку и белковые биомаркеры этой уязвимости. Поскольку таргетные терапевтические средства для конкретных типов рака могут давать тканеспецифические реакции, авторы провели отдельное исследование, чтобы идентифицировать ассоциации лекарство-белок на тканевом уровне; таких ассоциаций выявили 108.
Наконец¸авторы проверили, снижается ли прогностическая способность при исследовании меньшего количества белков. Оказалось, что выбор случайных подмножеств из 1500 белков из полного набора данных позволяет достичь 88% мощности прогнозирования реакции на лекарство по сравнению с полным набором. Это подчеркивает взаимосвязь и совместную регуляцию белковых сетей, отмечают исследователи.
Источник
Goncalves, E., et al. Pan-cancer proteomic map of 949 human cell lines. // Cancer Cell, published online 14 July 2022. DOI: 10.1016/j.ccell.2022.06.010


 Меню
Меню





 Все темы
Все темы



 0
0