Как прокрутить эволюцию быстрее
Геном растений полон загадок. Некоторые из них интересны с точки зрения фундаментальной науки, другие дают ключ к улучшению качества сельскохозяйственных культур. О том, почему для решения этих загадок лучше всего подходит нанопоровое секвенирование, рассказывает Илья Киров.

На конференции Oxford Nanopore Day 2022, которая состоялась 21 января 2022 г., большой интерес вызвал доклад старшего научного сотрудника ФГБНУ ВНИИСБ Ильи Кирова. Он рассказал о CANS (Cas9-targeted Nanopore sequencing) — технике нанопорового секвенирования, основанной на способности комплекса нуклеазы Cas9 с гайд РНК связываться с определенными последовательностями в геноме. Группа Ильи применила этот метод для поиска инсерций транспозонов в геноме арабидопсиса, а также для секвенирования генов глютенина у тритикале. Для работы исследователи используют платформу MinION. Редакция PCR.NEWS выяснила, для каких задач CANS подходит лучше всего, почему важно писать собственные программы для обработки результатов и в чем залог успеха командной работы.
 Арабидопсис. Credit: Илья Киров
Арабидопсис. Credit: Илья Киров
Фиксация момента
Как работает метод CANS?
В 2018 вышла статья в Nature Biotechnology, в которой Тимоти Гилпатрик с соавторами показали, что с помощью Cas9 можно направить секвенирование в определенное место генома. Оказалось, что метод достаточно прост. Если Cas9 смешать с гайд РНК и добавить получившийся рибонуклеопротеиновый комплекс к геномной ДНК, то он вполне себе пойдет и разрежет ДНК там, где нужно. Но здесь есть одна очень важная особенность: когда Cas9 разрезает ДНК, она на одном конце ДНК так и висит очень плотно. На втором конце остается фосфат. Естественно, если мы просто разрежем и пойдем секвенировать «Нанопором», адаптеры будут пришиваться везде, кроме того кусочка, где висит Cas9. Поэтому авторы статьи сделали очень простой трюк: они ввели в систему фермент, который дефосфорилирует концы. То есть ДНК на первом этапе подготовки дефосфорилируется, становится инертной для лигирования, к ее концам ничего нельзя пришить. Потом добавляют Cas9 к этой инертной ДНК, и после разрезания на одном из концов остается фосфат — в этом месте адаптер может лигироваться. После этого работа идет по обычному протоколу Oxford Nanopore.
У вас есть препринт, в котором описывается применение CANS для поиска вставок транспозонов у арабидопсиса.
Мы работали с мутантом арабидопсиса, в котором активны определенные мобильные элементы. Нам было интересно, куда они вставляются в ответ на разные стрессы. Обычно для этого секвенируют весь геном, но это дорого, если интересует конкретный мобильный элемент. Поэтому мы стали искать другие способы. Изначально метод CANS был придуман для того, чтобы секвенировать конкретные гены. Встал вопрос: куда направлять гидовые РНК для наших задач? Мы развернули гидовые РНК в обратную сторону от элемента. Таким образом, если какой-то мобильный элемент вставился в геном, гидовые РНК его найдут, и мы отсеквенируем участки, которые фланкируют этот элемент.
Почему вы выбрали метод CANS для решения этой задачи?
Мы хотели отслеживать не только инсерции, которые произошли у «папы» и «мамы» этого арабидопсиса, но и соматические мутации. Обычно, чтобы понять, куда вставился транспозон, нужно получать следующее поколение растений и заставлять транспозон наследоваться. Это очень непростая задача, потому что у растений, как и у человека, есть очень жесткая система, предохраняющая от наследования новых инсерций. Мы этого не хотели делать, нами двигала лень. Мы хотели активировать транспозон и посмотреть, куда он вставился, в этом же растении, не получая потомков. Вот какая была мечта. Метод CANS оказался очень чувствительным — он выхватывает соматические мутации. У мутантов арабидопсиса по 20–30 унаследованных инсерций определенного элемента, при этом метод CANS выявляет больше 800 соматических инсерций, и это значительно расширяет возможности для проверки разных гипотез.
Что еще важно здесь подчеркнуть: если мы хотим изучить, куда вставляется мобильный элемент, но исследуем инсерции в следующем поколении, мы не увидим инсерцию, которая попала в какой-то очень важный ген, и растение не выжило. Получается, что мы не видим реальной картины. На уровне популяций все еще сложнее. Тысячи лет накапливались эти инсерции, произошло достаточно жесткое давление отбора. С помощью метода CANS мы выявили инсерции, которые являются не результатом давления отбора, а следствием некой предрасположенности определенных участков ДНК к вставкам определенных мобильных элементов.
То есть вы фиксируете состояние генома в моменте?
Да. И это срез по всем клеткам.
Для обработки результатов секвенирования вы использовали собственный софт?
Да, мы использовали нашу программу NanoCasTE. Программа находит риды с фрагментами транспозона, она их «меряет», сравнивает с ожидаемым размером и потом говорит, где произошла инсерция.
Насколько вы доверяете белку Cas9? Много говорят о его нецелевой активности.
Офтаргеты есть. О некоторых мы знаем точно, мы их предсказали и видим их, когда обрабатываем результаты CANS. Возможно, менее специфичные офтаргеты мы не замечаем. Если есть различия между ложной мишенью и гайд РНК, теоретически Cas9 может в этом месте связаться, но его эффективности не хватает, чтобы дать нам риды.
Открытое комьюнити
На конференции у меня сложилось впечатление, что решения Oxford Nanopore не всегда работают из коробки. Поэтому исследователям приходится дорабатывать методы и разрабатывать свой софт, чтобы расширить применение нанопорового секвенирования. Это правда?
По поводу софта — правда, особенно при изучении растений. Не все алгоритмы, которые публикуются, можно использовать для растений. Их нужно как минимум дорабатывать и оптимизировать, а то и писать самому, что мы сделали для метода CANS. Нам пришлось написать софт, потому что его не было вообще. Реактивы для метода CANS, о котором я рассказывал на конференции, уже есть «в коробке» у Oxford Nanopore. Однако некоторые реагенты в Россию не так просто заказать, поэтому проще что-то сделать самим. Например, у нас есть белок Cas9, который необходим для этого протокола. Есть хорошая возможность на отечественных китах сделать гидовые РНК. Это получается намного дешевле.
Есть ли другие методы отслеживания вставок ретротранспозонов?
На самом деле их много. И для «Нанопора» есть вариант, который мы очень хотим попробовать: специфичное секвенирование на уровне прибора. Есть алгоритмы, которые связывают компьютер с каждой порой. И пора им сообщает сиквенс молекулы, которая проходит сквозь нее. Но если при обычном секвенировании все прочтения, грубо говоря, «складываются в папочку», то при таком методе сиквенс, который нарабатывается в режиме реального времени, сразу сравнивается с тем, что вы хотите увидеть. Например, вы хотите секвенировать транспозоны, для этого есть специальный набор программ. Они берут этот сиквенс, сравнивают с базой данных, которую вы подгрузили. Если сиквенс совпадает с базой данных, пора секвенирует. Но если сиквенс не тот, делается eject — пора «выплевывает» сиквенс назад. Это очень классно, потому что не нужен никакой Cas9. Но встает вопрос эффективности. На разных генах, на разных растениях методы работают лучше или хуже. Скорее, они комплементарны друг другу.
Существуют ли методы, не связанные с «Нанопором»?
Есть метод, основанный на вылавливании молекул ДНК с инсерциями, с помощью шариков, к которым пришиты олигонуклеотидные пробы. А затем происходит амплификация выловленных фрагментов и секвенирование короткими ридами. Это основной конкурент в детекции инсерций транспозонов, но минус его в том, что это дорого.
Надо сказать, что транспозоны любят вставляться в другие транспозоны. Если мы берем любой метод с короткими ридами, то сразу же возникает проблема: если транспозон вставился в какой-нибудь повторяющийся регион, сложно найти это место. Хоть в длинных ридах и много ошибок, они имеют улучшенную картируемость и однозначно говорят нам, где вставка.
Какие еще есть преимущества у «Нанопора»?
Самое главное, за что все любят «Нанопор», — кроме сиквенса он дает информацию о модификации ДНК. Это очень круто. У каждого метода есть плюсы и минусы, здесь нет однозначно «золотого стандарта». Если мы хотим выцепить инсерции и сразу же понять эпигенетику, то у «Нанопора» пока нет конкурентов. Мы готовим вторую версию статьи, которая выложена на bioRxiv. Добавится часть по эпигенетике, где мы показываем, как можно с помощью CANS увидеть профиль метилирования транспозона мутантного и не мутантного растений. Четко видно, что у растения дикого типа транспозон заметилирован очень жестко. А у мутанта, в котором этот транспозон активен, он прыгает, потому что нет метилирования. Сейчас мы пытаемся понять, что будет на уровне эпигенома на том месте, где произошла инсерция. С помощью CANS и с помощью нанопорового секвенирования это можно сделать.
Для определения эпигенетических модификаций тоже нужен специальный софт?
Да. По растениям это отдельная история, потому что у растений часто происходит метилирование цитозина в разных контекстах. Контекст — это две буковки, которые идут после цитозина. У растений есть все варианты контекста, где цитозин может быть метилирован, включая CHH и СHG (Н — это любая буковка, кроме G). Эта картина очень отличается от генома млекопитающих, где метилирование цитозина происходит в подавляющем большинстве случаев в контексте CG. Именно этого и ожидают алгоритмы, настроенные на данные человека, теряя при этом информацию о метилировании в не-CG областях. А у растений это важный регуляторный пласт, который управляет активностью транспозонов в том числе. К нашему счастью, буквально недавно, в 2021 году, вышла статья с новым алгоритмом DeepSignal-plant. Алгоритм очень хорошо распознает метилирование и натренирован именно на данных по растениям.
Биоинформатическая часть нанопорового сообщества открытая, люди обмениваются друг с другом кодами?
Да, это общепринято. Принцип большинства проектов в биологии — открытость. Особенно открытость кода, потому что это позволяет модифицировать программы. У «Нанопора» очень сильное комьюнити, в нем есть и «мокрые» биологи, и биоинформатики. На специальном сайте можно задать вопросы и посмотреть, что другие ответили. Это очень сильная сторона «Нанопора», потому что ты не один. Особенно это было важно на первых порах. Сейчас мы все реже обращаемся к сообществу. Мы первопроходцы, к нам уже обращаются, скорее. Под «Нанопор» разработка софта идет сногсшибательными темпами. В растительной биоинформатике, как и в человеческой, всегда есть огромное пространство для самостоятельного кодинга и самостоятельной работы с данными, потому что некоторые форматы уникальные, каждый геном уникален. Если ты делаешь что-то новое, нужно написать программу, потому что вручную обсчитывать данные было бы очень долго.
Вот простой пример с метилированием. Нам нужно было отобразить метилирование после программы DeepSignal-plant на каждом риде. Например, у меня 100 ридов, и какой-то сайт заметилирован на восьмидесяти, а другой сайт — на двух. Для этого нам нужно было написать свой скрипт, потому что готовых решений нет. Пока мы его писали, мне пришла на рецензию статья из хорошего математического журнала с уже известной программой по визуализации данных метилирования. И в программе нет варианта «визуализировать данные DeepSignal-plant». Либо ты ждешь и отстаешь, либо ты пишешь сам и идешь вперед. Это нормальный процесс.
На конференции некоторые спикеры упоминали, что для обработки данных нужен мощный компьютер.
Нужно понять, где будет происходить base calling. Если много данных, нужна видеокарта. У нас есть видеокарта nVidia TESLA V100, очень мощная, просто летает. Мы запускали один и тот же набор данных параллельно на двух серверах абсолютно одинаковой комплектации, с той только разницей, что на одном сервере есть видеокарта, и мы считали на ней, а другой сервер без видеокарты, и считали мы на CPU. Так вот, видеокарта посчитала за 20 минут, а на сервере без видеокарты будет считаться два дня. Ускорение сумасшедшее, особенно с метилированием, потому что при нем нужно сначала делать base calling нуклеотидов, а потом снова вызывать метилирование. Без видеокарты даже на геном арабидопсиса мы бы не сунулись.
Когда у нас видеокарта начала работать, первое время ко мне прибегали через день сотрудники и говорили, что в серверной что-то явно взлетает. У видеокарты своя система охлаждения, которая не дает ей нагреться выше 65°С. Первое время мы стояли под дверью серверной, чтобы ничего не случилось. Сейчас уже поспокойнее отслеживаем температуру. В общем, видеокарта — это важнейший элемент работы с «Нанопором».
Получается, что если мы хотим расширять применение «Нанопора», нам нужен не только сам секвенатор, но также дополнительные реагенты, дополнительный софт и хорошая видеокарта. Насколько это выгодно?
При нанопоровом секвенировании и большом объеме данных без этого никуда. Вообще, в науке сложно оперировать терминами «выгодно — невыгодно». Это не бизнес. В некоторых случаях может быть проще на аутсорс отдавать — сейчас в Москве есть места, где могут сделать нанопоровое секвенирование на хорошем уровне, и обсчеты могут сделать. Но в нашем случае «Нанопор» — основной инструмент. Мы занимаемся такой фракцией генома, где очень тяжело работать другими методами.
Вы ничего не отдаете на аутсорс?
На аутсорс есть смысл отдавать, когда, например, нужна другая ячейка. У MinION своя ячейка. Есть прибор PromethION, его ячейка содержит в несколько раз больше пор, но она в два раза дороже чем у MinION. Поэтому если нужно секвенировать с большим покрытием, то лучше отдать на аутсорс на PromethION, что мы и будем делать. Если есть небольшие эксперименты и небольшие геномы, как у арабидопсиса, то мы сами прекрасно это сделаем. Отсеквенировать 5–6 геномов мы можем на одной ячейке с достаточным покрытием для нас. Но 20–30 геномов лучше отдать на аутсорс, это будет дешевле.
 Тритикале. Credit: Илья Киров
Тритикале. Credit: Илья Киров
CANS и CATS
Почему в вашем препринте об инсерциях транспозонов в геноме арабидопсиса метод назван CANS, а в статье о генах глютенина у тритикале — nCAТS?
Изначально его назвали nCATS, гайд РНК были направлены внутрь гена. Нашу модификацию для мобильных элементов мы сократили, как нам было удобно — CANS.
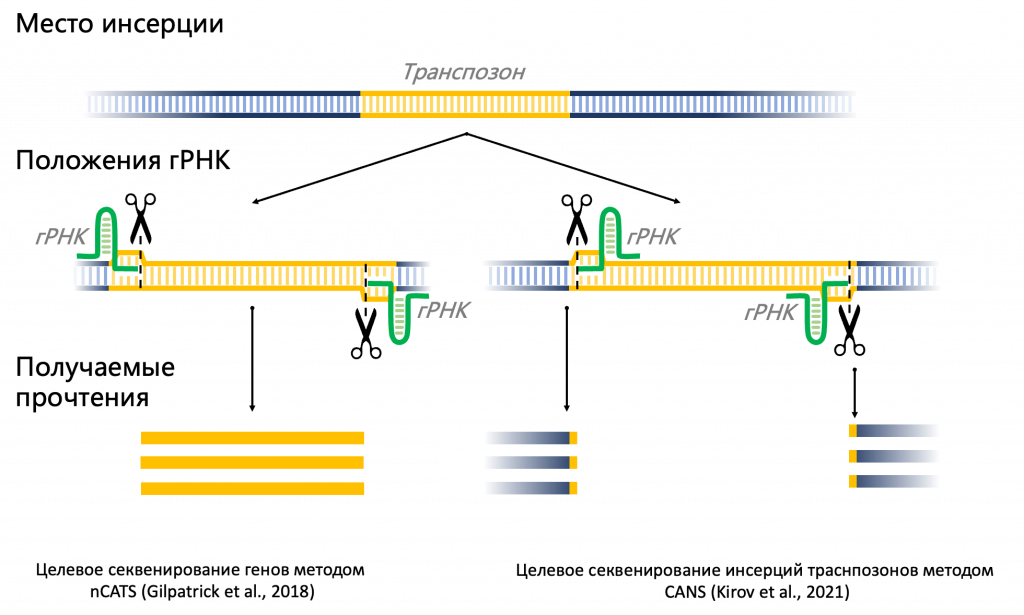 Схематическое изображение положений гидовых РНК и получаемых ридов при Cas9-опосредованном целевом нанопоровом секвенировании генов (слева) и инсерций траснпозонов (справа).
Схематическое изображение положений гидовых РНК и получаемых ридов при Cas9-опосредованном целевом нанопоровом секвенировании генов (слева) и инсерций траснпозонов (справа).
Имеет ли практическую значимость ваша работа по тритикале?
Да. Нужно понимать подоплеку. У тритикале гигантский геном. Он в восемь раз больше человеческого генома. Он собран плохо — собрали его недавно, и в сборке очень много ошибок. Мы изучали гены глютенина, это одни из самых практически ценных генов. Глютенины — это запасные белки, за которые мы ценим злаковые культуры. Они влияют на хлебопекарные качества. Казалось бы, эти гены должны быть собраны хорошо, но не тут-то было. Гены глютенина в геноме тритикале представлены отвратительно, они собраны совершенно неправильно. Дело в том, что эти гены на 80% состоят из маленьких повторов. В запасных белках порядок аминокислот не так важен, поэтому эволюционно они очень разнообразны. Нам нужно было отсеквенировать полный ген с промотором в наших линиях тритикале. Это практическая задача, потому что эти линии потом идут в селекцию. А гены — это мишени для селекции. Поэтому нам нужно было отсеквенировать ген, понять, какие методы есть, чтобы полностью покрывать вариабельность всего гена, а не отдельных его участков. Мы были вдохновлены именно этой идеей.
Была вторая идея, фундаментальная: понять, как связаны метилирование ДНК и экспрессия этих генов. Они экспрессируются на определенной стадии развития зерновки. Буквально две недели экспрессируются, и все. Мы можем немного управлять метилированием, и считается, что это может быть способом манипулирования качественными характеристиками зерна. Нам было важно понять, как заметилированы не отдельные участки генов, а гены полностью. Из методов подходит только нанопоровое севенирование. Мы благополучно с помощью nCATS нашли свой вариант гена для каждого из трех субгеномов тритикале и определили его метилирование. Мы увидели, что не только регуляторная область гена заметилирована, но и сам ген заметилирован, так называемое gene body methylation. К сожалению, мы больше не будем использовать nCATS для секвенирования генов тритикале, потому что мы потратили две ячейки MinION, чтобы получить покрытие генов на уровне десяти и более ридов. Этого было достаточно для наших целей, но этого мало. Две ячейки MinION — это экономически не целесообразно. Мы сейчас ищем альтернативные варианты, тоже с использованием «Нанопора», но без nCATS.
На темной стороне
Над чем вы сейчас работаете?
Первого февраля 2021 года была образована наша группа биологии мобилома растений во Всероссийском научно-исследовательском институте сельскохозяйственной биотехнологии. Так что мы отмечали первый день рождения.
 Группа биологии мобилома растений ВНИИСБ.
Группа биологии мобилома растений ВНИИСБ.
Мы изучаем «темную сторону» генома и сейчас пытаемся понять, как эти мобильные элементы вовлечены в создание разнообразия на Земле. Мы не просто смотрим, как геномы защищаются от мобильных элементов, — это мы знаем на достаточно хорошем уровне. Мы пытаемся изучить механизмы и биологию мобильных элементов, хотим узнать, как проходил их жизненный цикл, с какими компонентами клетки они взаимодействуют в этом жизненном цикле. Наша задача — выяснить, как самый многочисленный компонент геномной экосистемы взаимодействуем с остальной клеткой. Это практически важное направление, потому что оно позволит нам использовать мобильные элементы для создания новых генотипов растений. Сейчас мы делаем это на арабидопсисе. Благодаря нашим коллегам из Франции мы получили протоколы, которые позволяют запустить мобильные элементы и даже их закрепить в популяции. Результаты войдут во вторую версию препринта о CANS. Это стык нанопорового секвенирования и «мобилома».
В процессе эволюции, например, у лука или томата много инсерций мобильных элементов было накоплено за пять тысяч лет. Это был очень медленный процесс, но те инсерции, которые закрепились, ассоциируются с важными фенотипическими признаками. Томат — шикарный пример, у него инсерции определяют и форму плода, и цвет, и форму листа. Изучение биологии мобильных элементов позволит понять, можем ли мы эту эволюцию прокрутить быстрее. И здесь у нас в руках есть все инструменты.
Сколько сейчас человек в команде?
У нас девять человек.
А сколько нужно для успешной работы?
Нужны «всего лишь» умные и исполнительные ребята, которые желают работать. У нас все такие, других у нас нет. Мы же в науке добываем знания, мы же ищем все время, а если искать спустя рукава, ничего не найдешь. Поэтому у нас только те ребята, которые реально хотят что-то найти, с горящими глазами. Мы пытаемся что-то найти, и что-то находим, видите.
Компания SkyGen — дистрибьютор продукции Oxford Nanopore в России — считает, что научные знания должны быть доступны всегда.


 Меню
Меню





 Все темы
Все темы




 0
0