Опубликован первый клеточный атлас мозга млекопитающего
Проект BRAIN Initiative Cell Census Network Национальных институтов здравоохранения США, в котором приняли участие сотни исследователей, представляет клеточный атлас мозга мыши. Этому достижению посвящены десять публикаций в журнале Nature. Атлас включает в себя точную пространственную локализацию типов клеток, отличающихся по транскриптомным и эпигеномным характеристикам, описывает взаимодействие между клеточными типами в разных отделах мозга и позволяет сравнить систему регуляции экспрессии генов в мозге мыши, человека и других животных.
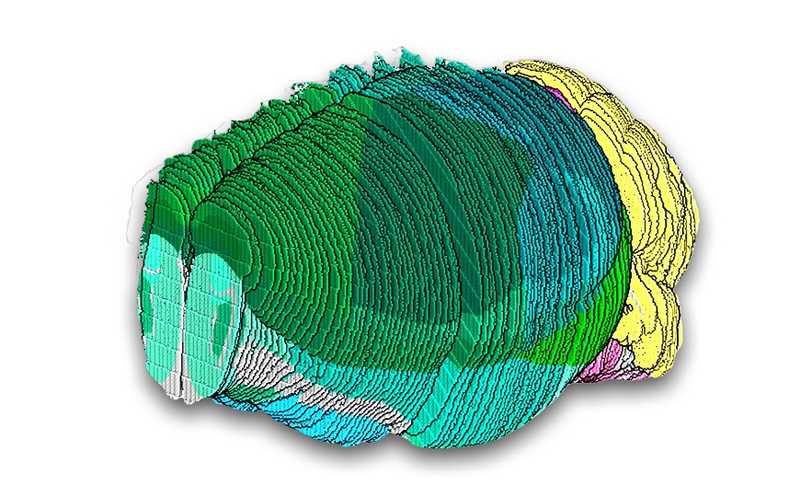
В недавнем выпуске журнала Nature опубликованы десять статей, посвященных клеточному атласу мозга мыши. Это первый подобный атлас мозга млекопитающего, результат многолетней работы сотен ученых, проведенной в рамках проекта BICCN (BRAIN Initiative Cell Census Network) Национальных институтов здравоохранения США. В задачи проекта входило построение сети клеток мозга мыши с классификацией по подтипам, точной локализацией и отслеживанием изменений в течение жизни. Атлас включил транскриптомные, эпигеномные и пространственные характеристики более 32 миллионов клеток во всех областях мозга мыши.
Основные методы исследования, которые при этом использовались, — это секвенирование РНК единичных клеток (scRNA-seq) и единичных клеточных ядер (snRNA-seq), методы пространственной транскриптомики, сочетающие исследование транскриптомов с точной локализацией, и анализ эпигеномов отдельных клеток. В общей сложности в мозге мыши были описаны более 5300 клеточных кластеров и более 8000 генов, которые служат маркерами различных клеточных типов. Работа дает более полное представление о том, как разные клеточные типы взаимодействуют друг с другом при помощи нейромедиаторов и нейропептидов, как работает иерархическая система клеточных типов для обеспечения работы целого мозга. Атлас был создан усилиями отдельных научных групп, результаты работы которых представлены в этих статьях.
В работе, проведенной под руководством специалистов Института наук о мозге Аллена, были интегрированы около 7 миллионов профилей scRNA-seq (около 4 млн прошли контроль качества) и результаты анализ около 4,3 миллионов клеток методом флуоресцентной гибридизации in situ (MERFISH). В результате были выделены 34 класса, 338 подклассов, 1201 супертип и 5322 кластера клеток. Для визуализации использовали ранее созданную онлайн-платформу Allen Brain Cell Atlas.
По словам Хункуй Зенг, соруководителя работы и директора Института Аллена, одно из основных открытий, сделанных при создании атласа, — это глубокая связь между геномной идентичностью клеток и их локализацией. Эта связь показывает, как пространственное расположение влияет на функции мозга, открывает путь к исследованию его эволюции и демонстрирует, как организовано взаимодействие между разными областями мозга.
Интересно, что ученые обнаружили разную клеточную организацию между дорзальными (верхними) и вентральными (нижними) отделами мозга. Более древние вентральные отделы представляют собой мозаику из разнообразных клеток, а более молодые дорзальные отделы включают меньше клеточных типов, при этом они более специализированы. По мнению авторов, это отражает тот факт, что нижние отделы несут базисные для выживания функции, а верхние важны для адаптации.
Авторы описали «коды», определяющие клеточную идентичность, которые включают транскрипционные факторы. Они также предложили инструменты, позволяющие генетически таргетировать отдельные типы клеток для изучения их функций и роли в развитии заболеваний.
«Мы знаем, что многие заболевания происходят из определенных областей мозга и, вероятно, определенных типов клеток, — говорит Зенг. — Имея в руках карту мозга, мы можем более пристально взглянуть на нарушения, происходящие при заболевании, а затем создать генетические или фармакологические средства, воздействующие именно на эти типы клеток для повышения эффективности и снижения побочных эффектов».
Институт Аллена также участвовал в создании детальной карты нейронов, которые связывают головной мозг со спинным. Используя мечение нейронов вирусными векторами и snRNA-seq, авторы разработали анатомический и транскриптомный атлас нейронов, дающих проекции в спинной мозг (SPN), относящихся к коре, гипоталамусу, среднему мозгу, ретикулярной формации ствола мозга и ядрам мозжечка. Эти нейроны образуют три раздела, 13 подклассов и 76 типов, обладающих уникальными транскриптомными и анатомическими свойствами. Первый раздел включает исключительно кортикоспинальные, руброспинальные и цереброспинальные возбуждающие нейроны, относительно гомогенные по транскриптому. Второй — гетерогенные возбуждающие и тормозные ретикулоспинальные нейроны. Третий — модулирующие SPN, экспрессирующие медленнодействующие нейротрансмиттеры и (или) нейропептиды и распределенные по гипоталамусу, среднему мозгу и стволу. Путем интеграции молекулярных характеристик и локализации нейронов в единый атлас, ученые продвинулись в понимании того, как эта сложная нейронная сеть контролирует функции организма и движения.
«Имея базовую карту этих клеточных типов, мы теперь можем изучать, как они изменяются при повреждении спинного мозга или инсульте, чтобы развивать таргетную терапию», — говорит Карла Винтер, соавтор исследования из Гарвардского университета.
В работе под руководством специалистов Медицинской школы Калифорнийского университета в Сан-Диего исследователи сравнили регулировку генной активности в мозге разных видов млекопитающих. Авторы проанализировали ее в первичной моторной коре человека, макаки, мармозетки и мыши, использовав мультиомиксный подход на уровне отдельных клеток. На 200 тысячах клеток они проанализировали экспрессию генов, доступность хроматина, ДНК-метилом и конформацию хромосом. Оказалось, что специфичный для клеточных типов паттерн генной экспрессии эволюционирует намного быстрее, чем паттерн экспрессии, общий для всех типов клеток.
«ДНК клетки подобна ее языку, — сказал Бинг Рен, ведущий автор статьи, профессор Медицинской школы Калифорнийского университета в Сан-Диего. — Подобно тому, как существуют определенные основные слова, которые являются общими для многих языков, существуют определенные гены и паттерны экспрессии генов, которые сохраняются у разных видов. Если мы научимся понимать и интерпретировать молекулярный язык мозга, это поможет нам узнать больше о работе мозга в целом и о том, что происходит в мозге при нейропсихиатрических условиях».
«Эволюция человека продолжалась миллионы лет, и большую часть этой эволюционной истории человек разделял с другими видами, — объясняет Джозеф Эскер, профессор Института Солка, соруководитель этой работы. — Только лишь данные по человеку не дадут нам достаточно информации для понимания того, как работает мозг. Заполненив бреши другими видами, можно получить ответы на многие вопросы и улучшить модели машинного обучения, предоставив им больше данных».
В другой работе ученые Калифорнийского университета в Сан-Диего составили атлас кандидатных цис-регуляторных элементов ДНК (cCRE) в мозге взрослой мыши. Для этого они проанализировали доступность хроматина в 2,3 млн клеток на 117 срезах мозга. Атлас включает около 1 млн cCRE и информацию о доступности хроматина в 1482 разных клеточных популяциях. Ученые применили искусственный интеллект для того, чтобы по последовательности ДНК предсказывать активность регуляторных элементов в отдельных типах клеток. Кроме того, они выяснили, что многие генетические программы, определяющие клеточные типы, находятся в областях генома, ранее ассоциированных с разными заболеваниями, такими как рассеянный склероз, анорексия и другие.
Специалисты Института биологических исследований Солка и их коллеги работали над созданием атласа метиломов мозга мыши. Они описали 301 626 метиломных профилей и 176 000 совместных профилей метилома — конформации хроматина 117 срезов из различных областей мозга; разработали классификацию клеток на основе метиломов, включающую 4673 клеточные группы и 274 подкласса; выделили 2,6 млн дифференциально метилированных областей. На основе метилома построили регуляторную сеть мозга, включающую транскрипционные факторы, регуляторные элементы и их потенциальные гены-мишени.
Исследуя молекулярную архитектонику мозга мыши, проанализировав около 6 миллионов snRNA-seq профилей, ученые использовали технологию пространственной транскриптомики, обратив особое внимание на более древние структуры мозга — ствол, средний мозг, продолговатый мозг, гипоталамус.
«Мы ожидали найти наибольшее клеточное разнообразие в этих областях, поэтому отдали приоритет исследованию их клеточных профилей, — говорит Эван Макоско, соруководитель работы, профессор Института Бродов. — В этих базисных областях сосредоточено много основных функций, в то же время им уделяется гораздо меньше внимания, чем коре. Наши результаты подчеркивает необходимость более глубокого их изучения».
Предположение подтвердилось — в эволюционно более древних частях мозга было найдено наибольшее разнообразие клеточных типов. Авторы также определили минимальный набор генов, определяющий уникальность клеточного типа.
Атлас мозга мыши — первый шаг для создания в будущем столь же детального атласа других видов, а в конце концов и человека.
Источники:
Zizhen Yao, et al. A high-resolution transcriptomic and spatial atlas of cell types in the whole mouse brain // Nature volume 624, pages 317–332 (2023) DOI: 10.1038/s41586-023-06812-z
Meng Zhang, et al. Molecularly defined and spatially resolved cell atlas of the whole mouse brain // Nature volume 624, pages 343–354 (2023) DOI: 10.1038/s41586-023-06808-9
Jonah Langlieb, et al. The molecular cytoarchitecture of the adult mouse brain // Nature volume 624, pages 333–342 (2023) DOI: 10.1038/s41586-023-06818-7
Shi H., et al. Spatial atlas of the mouse central nervous system at molecular resolution // Nature 622, 552–561 (2023). DOI: 10.1038/s41586-023-06569-5
Liu H., et al. Single-cell DNA methylome and 3D multi-omic atlas of the adult mouse brain // Nature 624, 366–377 (2023). DOI: 10.1038/s41586-023-06805-y
Zu S., et al. Single-cell analysis of chromatin accessibility in the adult mouse brain // Nature 624, 378–389 (2023). DOI: 10.1038/s41586-023-06824-9
Zhou J., et al. Brain-wide correspondence of neuronal epigenomics and distant projections // Nature 624, 355–365 (2023). DOI: 10.1038/s41586-023-06823-w
Zemke N.R., et al. Conserved and divergent gene regulatory programs of the mammalian neocortex // Nature 624, 390–402 (2023). DOI: 10.1038/s41586-023-06819-6
Winter C.C., et al. A transcriptomic taxonomy of mouse brain-wide spinal projecting neurons // Nature 624, 403–414 (2023). DOI: 10.1038/s41586-023-06817-8
Hahn J., et al. Evolution of neuronal cell classes and types in the vertebrate retina // Nature 624, 415–424 (2023). DOI: 10.1038/s41586-023-06638-9
Цитаты по пресс-релизам:
https://www.eurekalert.org/news-releases/1010700
https://www.eurekalert.org/news-releases/1010490
https://www.eurekalert.org/news-releases/1010965
https://www.nature.com/immersive/d42859 … index.html


 Меню
Меню





 Все темы
Все темы



 0
0