Рандомизация генома клеток человека — новый метод создания структурных перестроек
Ученые из США и Великобритании использовали технологию праймированного редактирования для создания клеточных линий с крупномасштабными генетическими перестройками. Они внедрили тысячи симметричных сайтов рекомбинации loxP в последовательности ретротранспозонов семейства LINE-1. По этим сайтам авторы запускали перестройки генома с помощью рекомбиназы, создавая сотни различных структурных изменений в каждой клетке. Такой подход позволил масштабно проанализировать, как структурные изменения в геноме влияют на характеристики клеток (их жизнеспособность и профиль экспрессии). Авторы предлагают использовать его для изучения генетической основы патологий и для разработки минимального генома млекопитающих.
Лишь около 1% генома человека кодирует белки, а функции некодирующих участков остаются во многом неизученными. Существенную долю генома — около 54% — составляют повторы. Для того, чтобы их охарактеризовать, ученые из Великобритании и США получили клетки человека с рандомизированным геномом и проверили на этой модели, как те или иные некодирующие области регулируют экспрессию генов и влияют на жизнеспособность клеток.
Для массовой рекомбинации исследователи применили праймированное редактирование, с помощью которого они встроили тысячи сайтов узнавания рекомбиназы в повторяющиеся последовательности ДНК.
На первом этапе авторы внесли симметричные loxP-сайты (loxPsym) в повторы LINE-1. Опыты проводили на клетках HEK293T и HAP1. Доля успешного редактирования составила около 1%, однако выживаемость клеток была низкой. Тогда исследователи решили контролировать редактирование с помощью доксициклин-индуцибельной системы. Клетки трансдуцировали лентивирусами, кодирующими целевой конструкт (гидовую РНК, направляющую праймированное редактирование LINE-1). Культивация полученных клеток в присутствии доксициклина и последующая селекция позволили увеличить долю отредактированных клеток — в конечном счете ученые добились редактирования около 8% всех LINE-1 в клетках HEK293T.
За несколько этапов индукции доксициклином и субклонирования (отбора наиболее перспективных отредактированных клеток) исследователи получили клон HEK293T, в котором было отредактировано 16% всех доступных участков LINE-1. Получение этого клона заняло год.
Также ученые попробовали применить данных подход к индуцированным плюрипотентным стволовым клеткам (иПСК) и фибробластам человека для оценки эффективности данной стратегии в доброкачественных диплоидных клетках. Для обоих типов клеток секвенирование выявило лишь минимальную вставку сайтов loxP (0,0028% для иПСК и 0,0036% для популяции фибробластов).
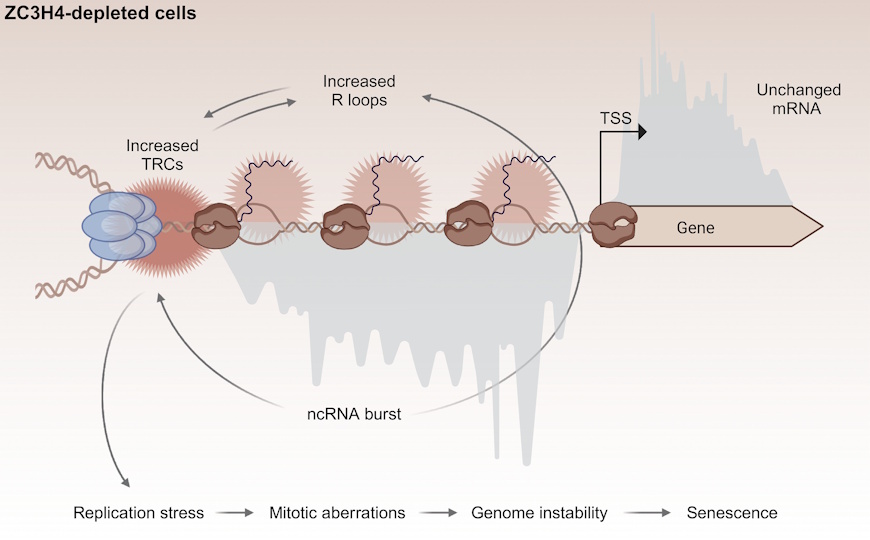
Чтобы точно определить расположение и количество интегрированных сайтов loxPsym, а также непреднамеренные изменения генома, ученые провели полногеномное секвенирование трёх отредактированных клонов. Они выявили 301 клональную вставку в HAP1, а в двух клонах HEK293T обнаружилось 638 и 1697 сайтов. Вставки loxPsym распределялись по геному достаточно равномерно — в среднем 14,3 вставки на хромосому (от 1 до 34) в клетках HAP1, — и демонстрировали сильную корреляцию с количеством LINE-1.
После внедрения этих вставок учёные обрабатывали клетки рекомбиназой TAT-Cre и анализировали изменения генома методом секвенирования с длинными прочтениями. Количество перестроек зависело от числа сайтов loxPsym на хромосомах, и в целом для клеток HAP1 было обнаружено 6 481 изменений, а для HEK293T — 1 166. Перестройки включали инверсии, делеции и образование экстрахромосомной ДНК. Наиболее часто встречалась конъюгация «в себе» (fold backs) — разновидность перестройки, создающая зеркальные инверсии, особенно в гомологичных хромосомах. Расстояние между сайтами loxPsym обратно коррелировало с вероятностью рекомбинации, причем влияние на эффективность рекомбинации оказывала как линейная, так и пространственная близость.
В процессе «перемешивания» геномов клеток были получены новые линии, различающиеся только по структурным вариантам. Для 29 таких клонов авторы провели полногеномное секвенирование всего генома, для 17 — секвенирование РНК. Всего они обнаружили 32 варианта перестроек, в том числе 15 делеций и 14 инверсий, а также две сбалансированные транслокации и одну изохромосому (две копии одного плеча хромосомы, соединенные центромерой). Во всех клонах было удалено 10 Мб последовательности (в среднем 345 Кб на клон), а 229 Мб — инвертировано (в среднем 7,9 Мб на клон). Изучив несколько клонов, авторы обнаружили, что изменения в экспрессии в основном связаны с потерей или изменением структуры генов в результате крупных делеций. Например, в одном из клонов HAP1 делеция 599 Кб привела к снижению экспрессии гена EPHA7, а делеция 742 Кб в другом повлияла на гены OLA1 и SP3, снижая жизнеспособность клеток.
В случаях, когда крупная делеция не затрагивала структуру гена, она, как правило, не влияла на его экспрессию. Однако в одном из клонов HEK293T наблюдались большие потери хромосом, что указывает на проблемы с митозом после создания дицентричных хромосом. В целом, авторы показали, что структурные варианты могут непосредственно влиять на экспрессию генов через изменения копийности или структуры экзонов, но редки случаи влияния через дистанционные взаимодействия или организацию хромосом.
Таким образом, рандомизация генома открывает возможности для воздействия на большое количество генов и некодирующих участков, чтобы исследовать более широкий спектр мутаций по сравнению с однонуклеотидными вариантами. Этот подход позволяет создавать новые клеточные линии с улучшенными характеристиками или оптимизированными свойствами, давая ценную информацию о связи между генотипом и фенотипом. Методику можно применять для поиска причин устойчивости раковых клеток к препаратам, а также проведения полногеномных анализов роли тех или иных последовательностей, что поможет интерпретировать патогенные варианты и глубже понять структуру и функции генома. Эти подходы также могут стать основой для разработки минимальных геномов млекопитающих.
Праймированное редактирование позволило провести системный скрининг мутаций TP53
Источник
Koeppel J. et al. Randomizing the human genome by engineering recombination between repeat elements // Science, Published: 30 January 2025. DOI: 10.1126/science.ado3979


 Меню
Меню





 Все темы
Все темы




 0
0