Компактная, но эффективная Cas9 обнаружена у стрептококка вымени
Для расширения спектра мишеней CRISPR-Cas систем, применяемых в науке и медицине, важно разнообразие их смежных с протоспейсером мотивов (PAM). Ученые из США обнаружили у бактерии Streptococcus uberis — патогена коров, который считается безопасным для человека, — ранее не описанный вариант Cas9. Ее PAM оказался богат AT, в отличие от GC-богатых PAM наиболее популярных Cas9. Сама нуклеаза демонстрировала уровень активности, сопоставимый с наиболее часто используемыми вариантами Cas9 от Staphylococcus aureus и Streptococcus pyogenes, но была меньше по размерам.

Открытие CRISPR-Cas систем произвело революцию в молекулярных методах, создав перспективные методы редактирования генома. Дальнейшая разработка dCas9, лишенных нуклеазной активности, расширила возможности системы и позволила регулировать экспрессию генов за счет связывания dCas9 с промоторами или энхансерами. Одно из ограничений CRISPR-Cas систем — специфичный для каждой Cas9 мотив, смежный с протоспейсером (protospacer adjacent motif; PAM). Эта последовательность из 2–6 нуклеотидов должна прилегать к мишени нуклеазы для ее успешной работы. Поэтому для расширения пула потенциальных мишеней редактирования необходимы ортологи Cas9 с различающимися PAM. Ученые из США обнаружили у лактобактерии Streptococcus uberis Cas9 систему, сравнимую по эффективности с традиционно используемыми системами, полученными от Staphylococcus aureus и Streptococcus pyogenes.
Авторы работы использовали пайплайн CRISPRdisco для поиска ортологов Cas9 у представителей лактобактерий (Lactobacillales). Эту группу бактерий выбрали из-за разнообразия и безопасности для человека. Для каждой предполагаемой Cas9, найденной в геномах этих бактерий, генерировали последовательность PAM и гидовой РНК, нацеленной на ген HBE. Всего для in vitro тестов отобрали 47 вариантов.
Отобранные и конвертированные в dCas9 последовательности, слитые с репрессорным доменом KRAB, интегрировали в лентивирусный вектор вместе с флуоресцентной меткой GFP, по которой оценивали экспрессию. Способность к эпигенетической регуляции проверяли на линии клеток K562, у которых целевой ген HBE
был помечен флуоресцентной меткой mCherry. Авторы начали тестирование именно с эпигенетической составляющей, так как работа dCas9 требует только успешного связывания с мишенью, но не нуклеазной активности — это может расширить круг последовательностей, которые могут быть пригодны к использованию. Из 47 нуклеаз удалось экспрессировать 34; из них пять эффективно подавляли экспрессию HBE. Специфичность работы трех наиболее эффективных dCas9 (полученных от S. uberis, S. gallolyticus и S. iniae) подтверждали транскриптомным анализом.
Нуклеазную активность Cas9 проверяли на линии клеток HEK293T, также используя HBE в качестве гена-мишени. По результатам секвенирования наибольшую эффективность редактирования продемонстрировала Cas9 S. uberis — она составила около 35%. В дополнительных тестах на гене TRAC (одна из терапевтических мишеней реакции «трансплантат против хозяина» при аллогенной терапии Т-клетками) эта нуклеаза показала сравнимую эффективность. Оптимизация длины спейсера гидовой РНК с 20 до 21 нуклеотида позволила повысить эффективность редактирования до 60%.
Cas9 S. uberis показала эффективность в редактировании отдельных оснований и эпигенетической активации генов, а также в подавлении экспрессии еще одной потенциальной терапевтической мишени — гена PCSK9, репрессия которого снижает уровень холестерина при гепатите.
Таким образом, исследование выявило еще одну перспективную Cas9 систему у бактерий S. uberis. Авторы отмечают, что PAM этой нуклеазы насыщен A/T, в противовес G/C-богатым PAM наиболее часто используемых Cas9 (таких как SpyCas9). Кроме того, Cas9 S. uberis отличается меньшими размерами, что упростит доставку системы редактирования в клетки. Эта и другие идентифицированные в работе нуклеазы расширяют спектр возможных мишеней генного и эпигенетического редактирования.


 Меню
Меню





 Все темы
Все темы
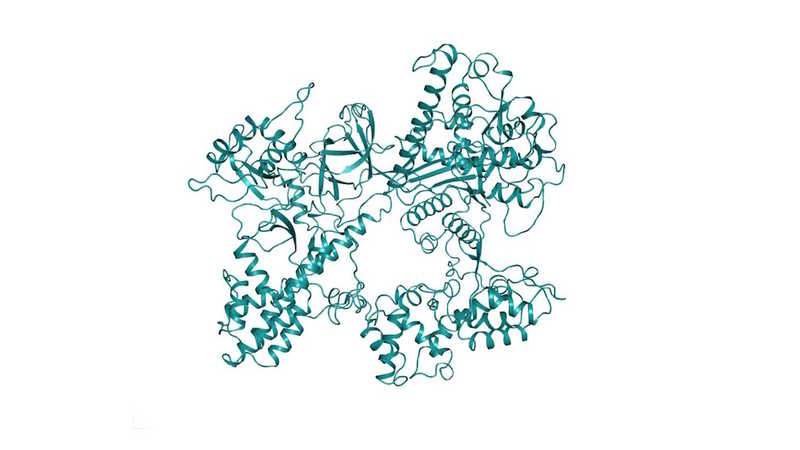 Предсказанная структура Cas9 S. uberis.
Предсказанная структура Cas9 S. uberis.



 0
0