Транскриптомное профилирование 1,3 млн. клеток мышиного мозга
Инструменты Chromium компании 10x Genomics позволяют провести транскриптомное профилирование миллионов клеток в одном эксперименте. Решения 10x набирают популярность за рубежом, но российские ученые мало знакомы с ними. PCR.NEWS представляет перевод рассказа специалистов компании о профилировании 1,3 млн. клеток мозга мышиных эмбрионов.
Credit: defun | 123rf.com
Технологии высокопроизводительного секвенирования РНК единичных клеток (scRNA-seq) используются для получения транскриптомных данных по всему геному и полностью изменили возможности в изучении сложных биологических систем. Однако большинство методов scRNA-seq рассчитаны на анализ от нескольких сотен до нескольких тысяч клеток за один эксперимент, что гораздо меньше количества клеток, входящих в состав большинства тканей и органов. Технологии анализа от сотен тысяч до миллионов клеток за один раз позволят существенно продвинуться в изучении сложных биологических систем.
Одна из таких технологий — Chromium Single Cell Gene Expression Solution от 10x Genomics. Результаты работы, выполненной с использованием этой технологии, представлены в Nature Communications. Chromium Single Cell Gene Expression Solution — это полностью интегрированный метод для цифрового профилирования 3’-концов мРНК, основанный на микрофлюидике. Метод позволяет охватить от сотен до миллионов клеток. В работе масштабируемость платформы и программного обеспечения 10x продемонстрировали на головном мозге млекопитающих — одной из самых сложных биологических систем в природе. Было проведено профилирование примерно 1,3 млн. клеток мозга, взятых из двух мышиных эмбрионов в возрасте 18 дней. За один эксперимент с помощью Chromium Single Cell Gene Expression Solution удалось выявить основные нейрональные и другие типы клеток, а также обнаружить множество подструктур в каждом из основных подтипов клеток мозга, включая редкие интернейроны. Обогащение клеток при этом не требовалось.
Ученые работали с образцами коры, гиппокампа и желудочковых областей головного мозга мышиных эмбрионов. Образцы были подготовлены согласно протоколу 10x Genomics для расщепления мышиной зародышевой нервной ткани. Детали протокола Chromium Single Cell Gene Expression доступны на сайте 10x Genomics. Баркодированные библиотеки кДНК были приготовлены с помощью реагентов из наборов Single Cell 3’ v2 (код продукта 120237). С помощью 17 чипов были приготовлены 133 библиотеки, в состав каждой из которых вошел материал из приблизительно 10 тысяч клеток.
Библиотеки были отсеквенированы с помощью проточных ячеек 11 HiSeq 4000 (Illumina) по протоколу парноконцевого секвенирования с конфигурациями запуска 26 пар оснований (R1), 8 пар оснований (i7) и 98 пар оснований (R2). Итоговая глубина секвенирования составила примерно 18 500 прочтений на клетку.
Анализ данных, включающий демультиплексирование, обработку баркодов, подсчет транскриптов и кластеризующий анализ, проводился с помощью программного обеспечения The Cell Ranger. Сначала данные по каждой библиотеке обработали по отдельности. Затем данные по всем 133 библиотекам объединили и получили сборную матрицу генов и баркодов. Наконец, объединенные данные обработали с помощью анализа главных компонент и кластерного анализа. Выравнивание, агрегация и подсчет транскриптов были выполнены на полном наборе данных. Для кластерного анализа использовали набор из случайно выбранных 20 000 клеток.
Далее выходные файлы программы Cell Ranger (в формате .cloupe) визуализировали с помощью Loupe Cell Browser (рис. 1). Это ПО для визуализации и анализа данных Chromium Single Cell 3’, подходящее для Windows и MacOS. В главном окне браузера данные представлены как две пространственные проекции tSNE. Инструменты позволяют пользователям выделять важные гены, определять типы клеток и исследовать подструктуры в кластерах клеток.
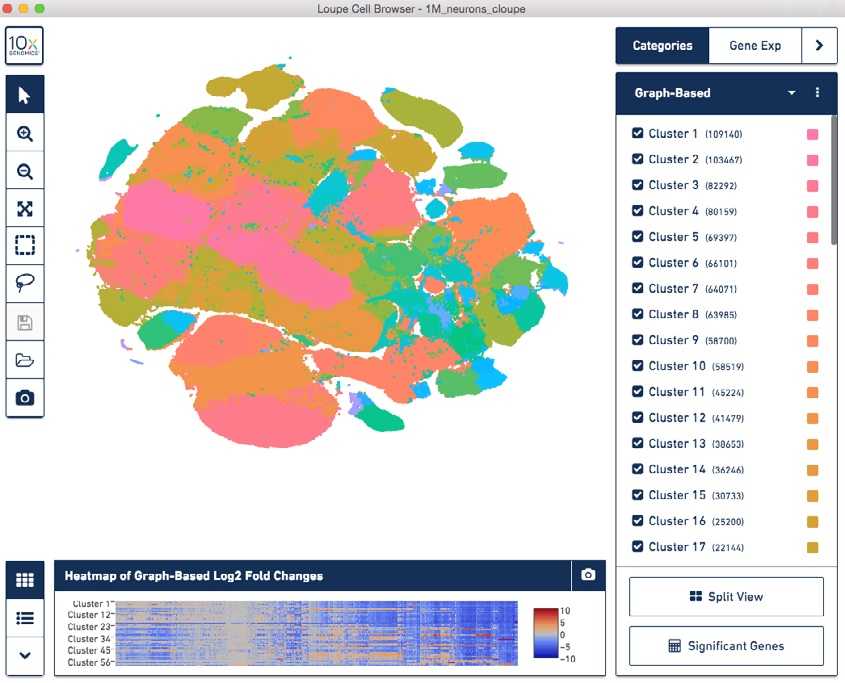 Рисунок 1. Скриншот окна программы Loupe Cell Browser, на котором отображены 1,3 млн. клеток мозга. Основные функции Loupe Cell Browser — динамическая интерактивная визуализация диаграмм рассеяния для tSNE, обобщение подгрупп, выбранных вручную, идентификация дифференциально экспрессируемых генов между группами, создание тепловых карт экспрессии генов, экспорт таблиц и графиков, содержащих данные по дифференциальной экспрессии генов.
Рисунок 1. Скриншот окна программы Loupe Cell Browser, на котором отображены 1,3 млн. клеток мозга. Основные функции Loupe Cell Browser — динамическая интерактивная визуализация диаграмм рассеяния для tSNE, обобщение подгрупп, выбранных вручную, идентификация дифференциально экспрессируемых генов между группами, создание тепловых карт экспрессии генов, экспорт таблиц и графиков, содержащих данные по дифференциальной экспрессии генов.
Менее 0,5% клеток несли >5% митохондриальных транскриптов, что свидетельствует о том, что большинство клеток находились в нормальном состоянии (рис. 2а). При глубине секвенирования порядка 18,5 тыс. прочтений на клетку в среднем для каждой клетки было идентифицировано около 1 870 генов и 5 000 транскриптов (рис. 2b и 2с).
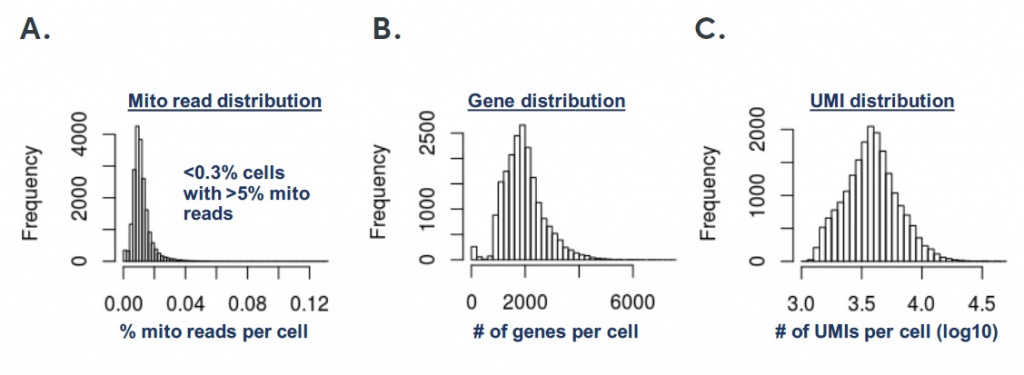 Рисунок 2. Анализ качества 20 000 клеток.
Рисунок 2. Анализ качества 20 000 клеток.
Доли нейрональных и других клеток можно оценить на основании данных по экспрессии маркерных генов в разных кластерах (рис. 3). Около 75% проанализированных клеток составляют нейроны. Примерно 60% из них приходится на возбуждающие нейроны и 15% — на тормозящие. Доля глиальных клеток составила около 25% всех клеток, из них примерно 12% приходится на астроциты и 9% — на олигодендроциты.
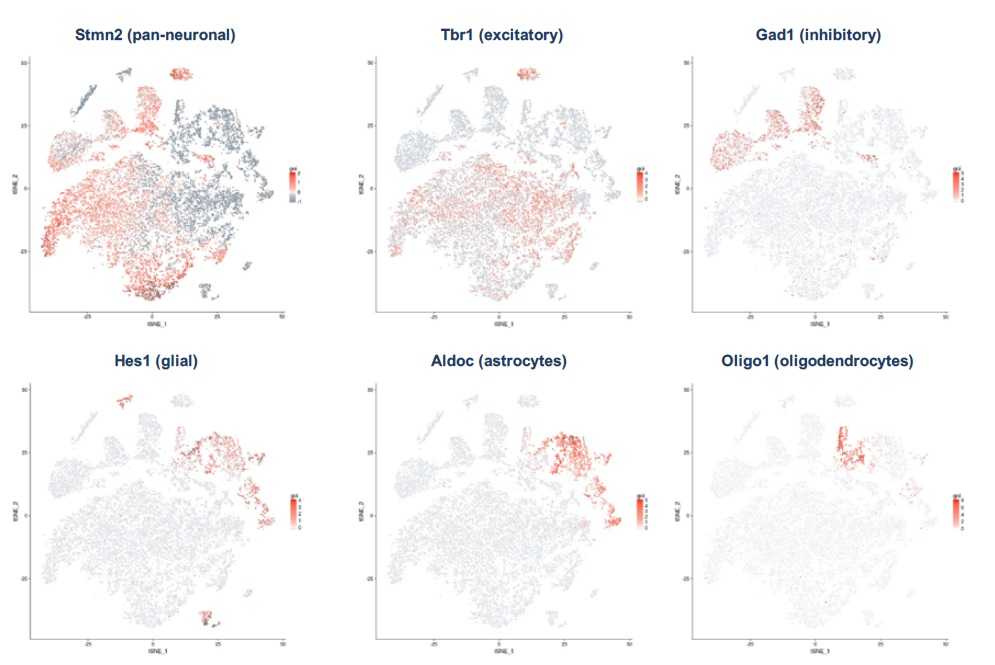 Рисунок 3. Профили экспрессии генов, наложенные на проекции tSNE, для шести выбранных маркерных генов клеток мозга.
Рисунок 3. Профили экспрессии генов, наложенные на проекции tSNE, для шести выбранных маркерных генов клеток мозга.
На основании экспрессии маркерных генов можно выделить семь основных классов клеток: возбуждающие и тормозящие нейроны, астроциты, муральные клетки, олигодендроциты, микроглиальные клетки и клетки эпендимы (рис. 4а). Клетки мозга также были классифицированы на основании наибольшего сходства их профилей экспрессии генов с референсными транскриптомами, полученными в Алленовском институте по изучению головного мозга (США) (рис. 4b). Обе классификации дали одинаковый результат. В обоих случаях паттерны кластеризации указывают на то, что все основные классы клеток имеют сложную внутреннюю структуру.
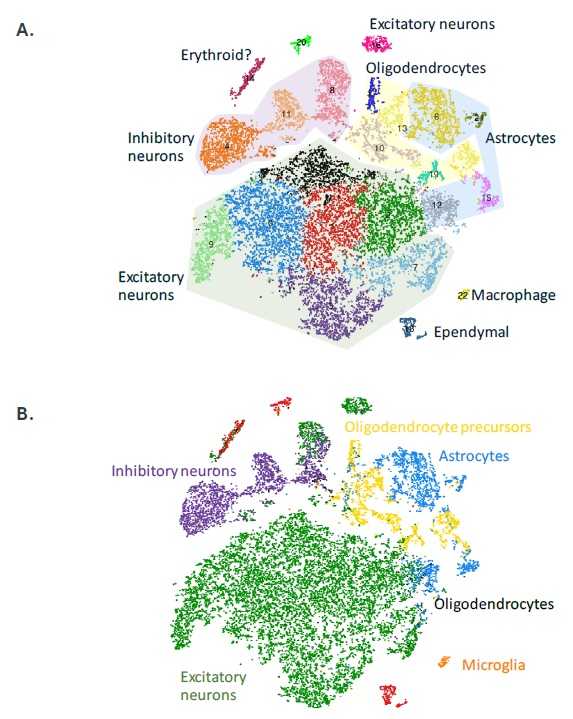 Рисунок 4. Проекции tSNE двадцати тысяч клеток мозга, в которых каждая клетка представлена точкой. (A) Штриховкой закрашены основные кластеры, выделенные в наборе данных, закрашивание цветом областей внутри заштрихованных зон дополнительно показывает сложность каждого из основных классов клеток мозга. (B) Клетки раскрашены в соответствии с их лучшим совпадением с общим профилем экспрессии генов в референсных транскриптомах (Tasic et al., 2016).
Рисунок 4. Проекции tSNE двадцати тысяч клеток мозга, в которых каждая клетка представлена точкой. (A) Штриховкой закрашены основные кластеры, выделенные в наборе данных, закрашивание цветом областей внутри заштрихованных зон дополнительно показывает сложность каждого из основных классов клеток мозга. (B) Клетки раскрашены в соответствии с их лучшим совпадением с общим профилем экспрессии генов в референсных транскриптомах (Tasic et al., 2016).
Компания 10X Genomics выкладывает в свободный доступ данные эксперимента и программное обеспечение в расчете на экспертную оценку научного сообщества. С помощью Loupe Cell Browser пользователи могут легко исследовать данные по экспрессии 3’-концов генов, полученные с помощью программы Cell Ranger. Сырые данные FASTQ также находятся в свободном доступе.
В представленной работе 10X Genomics получила крупнейший на данный момент набор данных scRNA-seq для клеток мозга. Хотя детально были исследованы лишь 1,5% клеток, транскриптомные данные для всего генома 1,3 миллионов клеток открыты для исследовательского сообщества и могут быть использованы как для создания новых методов, так и получения новых результатов.
Подробнее о продуктах 10x Genomics и их приложениях можно узнать у представителей компании SkyGen — эксклюзивного дистрибьютора 10x Genomics в России.
Источник
Ниже приведены полезные ссылки с информацией о решениях и ресурсах 10x:
Программное обеспечение и ресурсы
Параметры работы и системные требования ПО Cell Ranger
Партнерский материал


 Меню
Меню





 Все темы
Все темы




 0
0