Китайские секвенаторы, российский эталонный геном и сплайсинг в scRNA-seq
Найдены ли полноценные замены ушедшим с рынка брендам и приборам для секвенирования, когда в госреестре средств измерений появится отечественный эталонный геном, кто лучше конструирует Cas-редакторы — ИИ или человек, и многое другое на «Геномном секвенировании и редактировании» в РНИМУ.

В конце мая 2024 года в РНИМУ имени Н.И.Пирогова прошла Всероссийская научно-практическая конференция центров геномных исследований мирового уровня «Геномное секвенирование и редактирование».
Участников приветствовал ректор РНИМУ Сергей Лукьянов. Он напомнил о достижениях биолого-медицинских наук за последний год, которые не были бы возможны без NGS, в том числе о регистрации оригинального российского препарата против аутоимунного заболевания. Ведется работа над препаратом против диабета 1 типа, действие которого основано на том же принципе. Генная терапия еще недавно казалась чем-то далеким от практики, но сейчас в мире все больше препаратов получает разрешение к применению.
Однако в своем развитии генная терапия попадает в ловушку, отметил Сергей Лукьянов. Известно сотни заболеваний, при которых может помочь генотерапевтическое вмешательство, при этом один и тот же фенотип может вызываться множеством мутаций. С другой стороны, стоимость генотерапевтических лекарств будет колоссальной, если разрабатывать их в рамках КИ; речь может идти о сотнях миллионов рублей за дозу — бюджет целой больницы в небольшом населенном пункте. В России эту проблему помогает решить фонд «Круг добра». Но чем больше будет диагностироваться таких заболеваний и чем больше будет доступных препаратов, тем сильнее будет давление на здравоохранение, а отказать пациентам в медицинской помощи этически невозможно. Эта ситуация, если будет развиваться дальше, разрушит бюджет.
Решением может стать создание сети центров генной терапии, которые будут разрабатывать лекарство для конкретного человека, причем правила регистрации таких препаратов должны быть иными, чем у лекарств, предназначенных для массового применения. (Этот вопрос часто поднимает Денис Ребриков, который тоже присутствовал в зале.) Если такое же генетическое заболевание выявят у других людей, они смогут приехать в центр, где создано лекарство. При таком подходе стоимость препарата может составить 2–3 миллиона рублей, что уже приемлемо.
Платформы и приборы
На первой секции с докладами выступили разработчики и поставщики аппаратуры. Анастасия Бейзер (ООО «Аламед») рассказала о платформе NGS от китайской компании Cygnus.
Платформы Cygnus используют флуорогенное пиросеквенирование в запечатанных проточных ячейках. Это секвенирование путем синтеза: флуорофор отщепляется от нуклеотида в момент его встраивания в нить ДНК и излучает свет, таким образом, синтезируется нативная ДНК. Компания запатентовала свою технологию многократного запечатывания ячеек при помощи водно-органической фазы. Особенность технологии – одноцветная детекция, высокая скорость и отсутствие необходимость синхронной детекции всех ячеек, а также высокое качество данных — Q40 ˃80%. Качество Q40, что в 10 раз больше, чем у распространенных приборов Illumina, позволяет обнаруживать редкие мутации или уменьшать глубину прочтения и тем самым снижать стоимость анализа.
У Cygnus есть проточные ячейки разных типов, с системой совмещения различных адаптеров, что позволяет использовать библиотеки, подготовленные как для Illumina, так и для MGI.
Секвенатор Cygnus S100 обеспечивает пропускную способность 1 30 Гб, длины прочтений — SE75, PE150. Производитель предлагает специальные протоколы для быстрого и для точного чтения. Стоимость запуска ниже, чем у любого конкурента на рынке, включая GeneMind, подчеркнула докладчица.
Также Анастасия Бейзер рассказала о чиповой технологии цифровой ПЦР от китайской компании Turtle: полностью автоматизированная система, которая обеспечивает заполнение не менее 19 тысяч лунок из 20 тысяч и совместима с разными производителями мастер-миксов.
Доклад Надежды Волгиной (Диаэм) назывался «NGS для обнаружения низкочастотных мутаций. Поймай меня, если сможешь». Точность стандартного NGS не позволяет выявлять низкочастотные мутации, в том числе опухолевые, отметила она. В клональных опухолях все клетки имеют один и тот же геном, но так бывает не всегда: каждая клетка может иметь уникальный геном, отражающий мутационную сигнатуру родительской клетки. При использовании стандартных подходов сигналы единичных клеток неотличимы от шума. Решение этой задачи ценно не только с практической, но и с фундаментальной точки зрения, оно улучшает наше понимание эволюции опухолей. Существующие протоколы обычно ограничиваются митотически активными клетками, но как быть с терминально дифференцированными клетками, которые тоже могут содержать мутации?
Дуплексное секвенирование, то есть секвенирование обеих нитей ДНК, увеличивает точность в 100 000 раз по сравнению со стандартным NGS. Сравнение ридов, полученных с двух нитей (что обеспечивают специальные пары адаптеров), позволяет отличить истинные замены нуклеотидов от ошибок. (Дуплексное секвенирование исследователи из Института Сенгера в статье 2021 года назвали NanoSeq; число ошибок снижается до менее пяти на 109 пар оснований.)
Заменой Illumina, которая ушла с российского рынка, может стать китайская GeneMind, заметила докладчица. В частности, FASTAseq300 – аналог MiSeq, а GenoLab M — аналог NextSeq 2000.
По мнению Александра Богуна (ООО «Р-Ген производство»), нишу MiSeq способен занять и DNBSEQ-G-99 от MGI Tech. Докладчик напомнил, как выглядел рынок секвенаторов до того, как начались сложности с поставками. Большие приборы всегда обеспечивали более низкую стоимость секвенирования но у них была высокой стоимость запуска. Для таких задач, как анализ геномов патогенных микроорганизмов, привлекательным было полупроводниковое секвенирование, такие приборы, как Genexus компании Ion Torrent. Они обеспечивали высокую скорость проведения анализа, позволяя получить результат на следующий день. Когда их заменили приборами Illumina, скорость выдачи заключения замедлилась.
С точки зрения докладчика, имеющего значительный опыт исследований геномов микроорганизмов, идеальный секвенатор должен совмещать высокую скорость (результат в течение суток) с достаточной точностью, которая позволила бы снизить требования к кратности покрытия, и высокой производительностью. Желательна не слишком высокая стоимость запуска, чтобы не приходилось ждать, пока наберется достаточное количество образцов. Длина прочтений — 250РЕ и более, чтобы можно было использовать большинство панелей для таргетного обогащения на основе ПЦР без изменения протокола. Другие полезные опции — несколько ячеек, чтобы запускать несколько образцов одновременно, возможность срочного запуска анализа, возможность работы с разными наборами реагентов и техподдержка на территории РФ.
Этим требования удовлетворяет DNBSEQ-G-99, который докладчик назвал «супер-MiSeq». Скорость секвенирования сравнима с Genexus, причем пробоподготовка дешевле. Также у него теперь есть высокое качество прочтения, и это «единственная машина, которая хорошо поддержана нашем рынке». Круг применений этого прибора достаточно широк — он включает онкопанели, малые панели для наследственных заболеваний, полноэкзомное секвенирование, неинвазивные пренатальне тесты и преимплантационных генетический скрининг, секвенирование РНК.
Об опыте применения секвенаторов китайского производства для экзомного и таргетного секвенирования рассказала Анастасия Капуста (компания «Хеликон»). Два года назад отрасль в целом и молекулярная онкогенетика в том числе оказались в непростой ситуации, но «Хеликон» сумел найти решения для экзомного и таргетного секвенирования. Приборы MGI успешно работают на российском рынке. Среди решений для таргетного секвенирования — панели компании Nanodigmbio для секвенирования полного и клинического экзома. Помимо готовых онкопанелей, Nanodigmbio предлагает и кастомные, включающие мишени по выбору пользователя, причем дизайн можно сделать бесплатно. Продукцию Nanodigmbio высоко оценили сотрудники МГНЦ, Сеченовского университета и многие другие специалисты, подчеркнула докладчица.
Панели совместимы с приборами MGI линейки DNBSEQ, при этом наиболее широко представленные на рынке секвенаторы DNBSEQ-G50 и DNBSEQ-G400 имеют российские РУ. Высокопроизводительные приборы DNBSEQ T7 работают в российском проекте «100 000 геномов + я» (подробнее на PCR.NEWS). DNBSEQ T7 долгое время был самым производительным в линейке, но его превзошел DNBSEQ Т20, предназначенный для крупномасштабных популяционных исследований (72 Тб за запуск, или 200 экзомов человека за запуск, или 50 000 геномов человека в год).
Зав. лабораторией общей, молекулярной и популяционной генетики НМИЦ эндокринологии Минздрава РФ Сергей Попов познакомил слушателей с результатами сравнения двух технологий секвенирования РНК единичных клеток: scRNA-seq (10x Genomics) и scFact-seq (Seek One); поставщик — компания SkyGen. Анализировался переход клеток из одного состояния в другое (траектории экспрессии) при патологии коры надпочечников. Обе платформы работают сходным образом, однако преимущество scFact-seq — секвенируется полная длина транскриптов, а не только 3’-концы; также у нее существенно больше UMI count (абсолютное число транскриптов) на клетку. Есть преимущества и в параметрах секвенирования.
Докладчик отметил, что технология компании Seek One копирует 10х, но в плане анализа данных scFact-seq информации больше. Это касается в том числе анализа регулонов (взаимосвязи транскрипционных факторов с регуляторными областями генов): данные более представлены и более точны.
В следующем докладе также были представлены результаты решения одной и той же задачи средствами двух платформ, в данном случае принципиально различных. Ксения Климина (ФНКЦ ФХМ им. Ю.М.Лопухина ФМБА России) рассказала об изучении кишечной микробиоты мышей с помощью классического NGS (Illumina) и нанопорового секвенирования (Oxford Nanopore Technologies). Эта работа была частью исследования влияния биодобавок на микробиоту. Общеизвестно, что преимущество Illumina – высокая точность, а ONT — длинные прочтения. Результаты метагеномного анализа на этих двух платформах могут существенно различаться.
При анализе метагенома методом секвенирования можно прочитывать участки генов бактериальных 16S рРНК или же читать фрагменты всего генома. Докладчица с коллегами изучили, как влияют на результат по таксономическому разнообразию бактерий различные комбинации праймеров при секвенировании 16S рРНК на приборах Illumina и ONT. Также сравнили результаты полногеномного секвенирования.
При секвенировании 16S рРНК на Illumina читали регионы V3 и V4, на ONT — полная последовательность гена. Несмотря на схожее количество прочтений, сквенирование на Illumina выявило большее альфа-разнообразие. При этом Illumina обнаружила 73 рода, ONT — 86, и только 43 рода было выявлено с помощью обоих подходов. Интересно, что Bacteroides лучше определялись на Illumina, а Firmicutes — секвенированием на ONT. С другой стороны, между данными полногеномного секвенирования по двум платформам наблюдалась высокая корреляция. В то же время класс Bacilli лучше обнаруживался секвенированием гена 16S рРНК, чем полногеномным секвенированием.
Китайские наборы реагентов для секвенирования экзома человека с традиционными наборами от западных производителей сравнивала Вера Белова (РНИМУ им. Н.И. Пирогова). Протяженность всех экзонов в человеческом геноме — около 1%, при этом клиническая значимость полноэкзомного секвенирования всего на 2–6% ниже, чем у полногеномного (хотя ниже чувствительность к структурным вариациям и и вариациям числа копий).
Классические коммерческие наборы для экзомного обогащения производят Agilent, Roche, Illumina. Сейчас их заменили китайские производители — Vazyme, Nanodigmbio, MGI и др. Конечно, всех интересует, как скажется на результатах переход на зонды от китайских производителей. Докладчица привела ссылки на две публикации 2022 года. В одной сравнивается разработанный авторами протокол на базе зондов Agilent с решениями MGI, в другой — зонды Agilent Roche.
92% регионов таргетов — общие для наборов Agilent, Roche, Vazyme, Nanodigmbio, подчеркнула Вера Белова. Для сравнения исследовали стандартный геномный образец Е701, и результаты говорят о достаточно высоком качестве китайских продуктов, которые по отдельным параметрам (в частности, по полноте обогащения при том же количестве ридов) даже превосходят Agilent и Roche. «Можно переходить, многие коллеги уже перешли», — сказала докладчица в завершение.
Результаты
Затем начала работу секция Центров геномных исследований мирового уровня. Оптимизации системы хранения и обработки данных высокопроизводительного секвенирования был посвящен доклад Николая Кулемина (ФНКЦ ФХМ им.Ю.М. Лопухина ФМБА России). Это весьма нетривиальная задача. Необходимо организовать загрузку данных с секвенаторов; разработать систему управления загрузками и анализа данных; подобрать и адаптировать систему хранения данных для работы с результатами NGS, оптимизировать ПО для решения типовых задач и разработки новых протоколов анализа данных, и наконец, минимизировать затраты времени на управление высокопроизводительной системой.
Результаты NGS в основном находятся в FASTQ-GZ текстовых файлах объемом 1—10 Гб. Данные часто содержат технические повторы. Их можно разделить на «типовые» (часто встречающиеся задачи, такие как экзомное и геномное секвенирование, GWAS, анализ экспрессии и др.) и «исследовательские» (разработанные впервые новые методы, то, что, условно говоря, делается один раз)
Во многих странах есть требование хранить сырые файлы FASTQ, отметил докладчик, и это стоит соблюдать, хотя в РФ такого регламента нет. Результаты обычно хранятся в виде VSF/gVSF. Хранить ли что-то промежуточное и как быть с техническими повторами — это тоже вопросы, которые необходимо решать.
Существенная проблема хранения — избыточность для отказоустойчивости. Решением могут быть переносные носители, RAID-структуры, облачные хранилища (здесь речь идет об облачном ПО, без связи с внешним миром; используется CERN – открытая система управления облачными хранилищами, «локальное облако»).
Докладчик рассказал, как организованы хранение и доступ к данным, об упрощении хранения метаинформации (еще одна типичная проблема — люди забывают, что они делали с данными), организации вычислений, которая позволяет избежать перегрузки памяти и других сложностей. Примечательно, что запуск каждого шага возможен не только в консоли, но и в телеграм-боте.
Российских детальных регуляторных требований по хранению данных секвенирования пока нет, сказал Николай Кулемин, отвечая на вопрос. Согласно закону № 152, данные необходимо хранить на территории РФ, и это единственное требование, которое однозначно трактуется. По поводу остального «возможным разночтения», в частности, по поводу организации доступа к данным. Так, ООО «Биотехнологический Кампус», который реализует национальную генетическую инициативу «100 000 + я», считает, что нуклеотидные последовательности человека имеют высшую степень персональности. Однако докладчик считает приемлемым вариант с доступом после согласия, подписанного участником исследования.
Сегодня существуют три актуальные версии референсного генома человека: GRCh37/hg19 (2009), GRCh38/hg38 (2013) и новейшая T2T (telomere-to-telomere, 2021). О переводе биоинформатических решений в NGS-лаборатории на сборку генома человека hg38 рассказала Юлия Василиадис (РНИМУ им. Н.И. Пирогова). Сравнение результатов секвенирования с референсным геномом позволяет определить индивидуальные вариации (замены нуклеотидов, инделы, вариации числа копий). Таким образом можно обнаруживать варианты, ассоциированные с заболеваниями, и другие медицински значимые особенности.
Сборка hg38 новее hg19, в ней добавились alt-контиги, hla-локусы, центромерные последовательности, отметила докладчица. (ссылка). При использовании актуальной сборки для биоинформатической обработки данных полногеномного и полноэкзомного секвенирования среднее и медианное значения покрытия не изменились, но увеличилась полнота покрытия. Также они коллили меньше снипов и инделов.
Дмитрий Чудаков (РНИМУ им. Н.И. Пирогова, Институте биоорганической химии им. М.М. Шемякина и Ю.А. Овчинникова) сразу предупредил, что этот его доклад звучит в первый раз и посвящен достаточно сложному вопросу — новому методу анализа данных секвенирования РНК единичных клеток (scRNA-Seq) с учетом сплайсинга. Статья об этом методе, который авторы назвали SANSARA (Splicing-Aware scrNa-Seq AppRoAch) опубликована в виде препринта на biorXiv.
Современные аналитические подходы scRNA-Seq не различают сплайсированную и несплайсированную мРНК. С другой стороны, существует возможность определения «РНК-скорости» (RNA velocity) — соотношение сплайсированной и несплайсированной РНК для каждого гена дает информацию об экспрессии генов и состоянии клетки, «векторе» ее движения. Так, присутствие в клетке несплайсированной мРНК может говорить о ее переходном состоянии, характерном для таких динамических процессов, как эмбриогенез или регенерация тканей. Подсчет различных типов молекул РНК, в том числе сплайсированных и несплайсированных делают возможными уникальные молекулярные идентификаторы (UMI).
Однако в стабильных клеточных субпопуляциях также может накапливаться несплайсированная резервуары мРНК, которая позволяет быстро и независимо от трансляции инициировать продукцию белка. Поэтому наличие тех или иных несплайсированных транскриптов может быть характерным признаком субпопуляции клетки. Насколько известно докладчику и его коллегам, никто еще не пробовал использовать информацию о сплайсинге мРНК при построении UMAP репрезентации данных о клеточных популяциях, и они это сделали.
Исследователи охарактеризовали с помощью SANSARA субпопуляции CD4 + Т-клеток периферической крови. В частности, в субпопуляциях регуляторных Т-клеток периферической крови (Treg), Th1 и цитотоксических CD4+ Т-клеток удалось выявить гетерогенность, картина сплайсинга некоторых генов в них различалась.
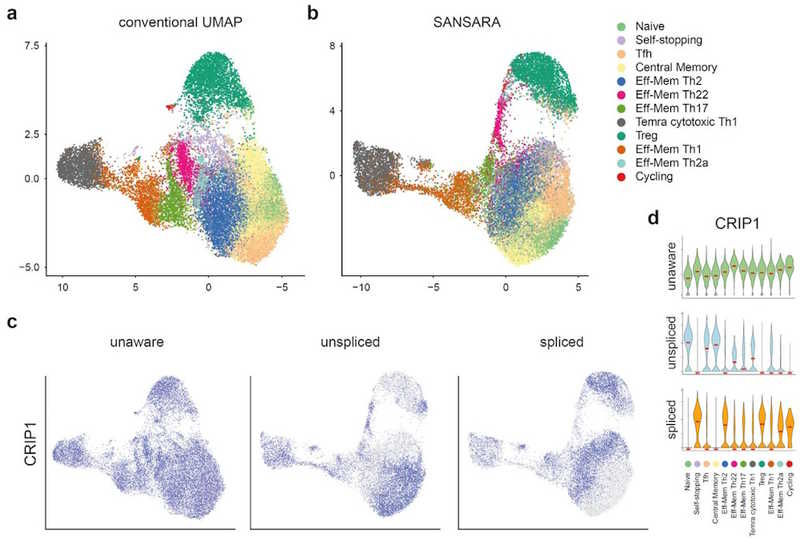
В транскриптомике появляется новое измерение, единичных клеток «возникает новая география», отметил докладчик. А так как scRNA-Seq — ключевой инструмент современной науки, это может оказаться очень важным.
Доклад Алины Самитовой (РНИМУ им. Н.И. Пирогова) назывался «Секвенирование экзома образцов эмбрионов человека, прошедших полногеномную амплификацию нового поколения». Примерно у 10% пациентов в программах вспомогательных репродуктивных технологий (ВРТ) не удается получить эмбрион, пригодный для переноса, и только 40% эмбрионов развиваются до стадии бластоцисты. Чтобы повысить вероятность успеха ВРТ и рождения здорового ребенка, проводятся молекулярно-генетические исследования: ПГТ-А (преимплантационное генетическое тестирования для выявления анеуплодий), ПГТ-М (для выявления мутаций).
Для исследования берут несколько клеток из трофэктодермы бластоцисты и проводят полногеномную амплификацию (WGA). Основные требования к ее продуктам — получение материала в достаточном количестве, высокий охват генома, однородность (равномерная представленность) амплифицируемого материала и неизмененная последовательность нуклеотидов.
Самый популярный метод амплификации — MDA (Multiple Displacement Amplification). Его преимущества — высокая точность применяемой для этого полимеразы phi29, широкая распространенность и простой протокол. Но есть и недостатки — неравномерная амплификация участков, возможность выпадения аллели. Для ПГТ-М этот метод не подходит.
Более равномерную амплификацию и покрытие генома дает метод PTA (Primary Template-directed Amplification). Сравнение MDA и PTA BioScryb, показало, что второй позволяет получить более короткие фрагменты дцДНК, и в итоге полнота покрытия экзома намного лучше: процент непокрытых регионов уменьшился до 1,6% с десятков процентов. Однако неравномерность данных в пуле сохраняется, и пока непонято, в чем причина, отметила докладчица.
Дмитрий Коростин (РНИМУ им. Н.И. Пирогова) рассказал о применении в реальной исследовательской практике первого российского стандарта генома человека E701 (того самого, который упоминала Вера Белова в своем докладе). О его получении мы писали в 2022 году.
Когда встает вопрос о качестве молекулярно-генетических исследований, необходимы стандарты, в том числе эталонные геномные последовательности. Существует Genome in a Bottle (GIAB), государственно-частно-академический консорциум, организованный Национальным институтом стандартов и технологий США для разработки технической инфраструктуры (справочные стандарты, эталонные методы, эталонные данные), поддерживающей внедрение геномного секвенирования в клиническую практику. (Подробнее о его работе на PCR.NEWS.) Однако доступ к эталонам GIAB сейчас осложнен.
Преимущество первого российского стандарта Е701 в том, что это геном мужчины, то есть он включает последовательности обеих половых хромосом. Образцы E701 прошли через полногеномное секвенирование с покрытием 30x в четырех независимых лабораториях Москвы. Участники консорциума — РНИМУ им. Н.И. Пирогова, НМИЦ АГП им. В.И. Кулакова, Курчатовский институт, НМИЦ радиологии. Ведется совместная работа с ВНИИ метрологической службы, Е701 находится на финишной прямой по внесению в госреестр средств измерений, отметил докладчик, и есть серьезные основания надеяться, что это произойдет уже летом 2024 года.

Диагностические и исследовательские лаборатории, которые занимаются секвенированием, отличаются производительностью, сроками процессинга и стандартизацией протоколов пробоподготовки и анализа. В диагностических лабораториях существуют жесткие стандарты, в исследовательских процесс может только создаваться. В любом случае для контроля многоэтапного протокола нужен стандартный геном.
На лабораторный процесс полногеномного секвенирования влияет очень многое: так, изменение температуры способно менять частоту представленности вариантов. Результат зависит от типов реагентов, оборудования, человеческого фактора и автоматизации; даже лот партии реагентов может играть роль. Поэтому так важно иметь стандарт.
Отвечая на вопросы, докладчик отметил, что источник генетического материала Е701 — культура фибробластов, хранящаяся в морозилке, как и у GIAB. Не предполагается делать более 10 пассажей, и, конечно, возможная контаминация или возникновение мутаций будут контролироваться секвенированием.
На секции «Редактирование генома и генотерапия» Дмитрий Карпов (ИМБ РАН) выступил с докладом «Разработка новых геномных редакторов: искусственный интеллект vs человеческий разум и удача». ИИ предложил структуру OpenCRISPR-1, которая более активна и специфична по сравнению с прототипом — SpCas9. (Подробнее о том, как она была создана, на PCR.NEWS.) На самом деле ИИ предложил огромное количество структур, но так как физически проверить можно было лишь небольшое количество — проверяли те, которые по структуре были наиболее похожи на известные Cas-редакторы. тем самым они избавились от белков, которые заведомо не работали. Однако у такого подхода есть ограничение — возможно, потенциал ИИ гораздо шире, чем у человека, но у человека нет возможностей это узнать. Также они выполнили инженерию направляющих РНК с помощью ИИ. Точность проверяли лишь на пяти сайтах, полногеномного сравнения не было, отметил докладчик.
Но и эта система, как и все популярные Cas-редакторы, работает по принципу «что не лечит, то калечит» — если не срабатывает гомологичная рекомбинация, за счет которой должна быть внесена высокоточная замена, мишень фактически уничтожена. Это не подходит для медицинских применений, если задача — не инактивация, а коррекция гена. Кроме того, OpenCRISPR-1 содержит замены в доминантных и субдоминантных эпитопах, которые могут вызывать сильный иммунный ответ; иммуногенность авторы пока не проверяли, отметил докладчик.
Затем Дмитрий Карпов перешел к достижениям «естественного» интеллекта. Он привел в пример публикацию 2023 года, авторы которой — Викаш Чаухан, Филипп Шарп и Роберт Лэнджер — показали, что структуры разрывов ДНК, созданные Cas9, определяют частоты различных путей репарации, от чего, в свою очередь, зависит точность. С учетом этого они разработали варианты Cas9 (vCas9), которые создают разрывы с липкими концами, «склонные» к точному редактированию. Возможности редактора они проверяли, превращая в культивированных клетках ген зеленого флуоресцентного белка GFP в ген синего флуоресцентного белка BFP. Возможно, это самый точный редактор на сегодняшний день.
Также докладчик рассказал о своей с коллегами разработке — редакторе iBolit24, также созданном на основе SpCas9. Его преимущество, важное для медицинских применений, — он практически лишен мутагенной активности, но редактирующая активность пока недостаточно высока (около одного процента). iBolit24, как и vCas9 Чаухана с коллегами, менее компактен, чем Cas9 и OpenCRISPR-1. Он также довольно значительно отличается от них по структуре.
В заключение Дмитрий Карпов сообщил, что International Journal of Molecular Science готовит спецвыпуск, посвященный CRISPR-Cas-системам и редактированию геномов.


 Меню
Меню





 Все темы
Все темы




 0
0